Способ идентификации генотипа и подтипа вируса гепатита c на биологическом микрочипе
Номер патента: 18102
Опубликовано: 30.05.2013
Авторы: Михайлович Владимир Михайлович, Изопет Жак, Заседателев Александр Сергеевич, Нико Флоранс, Грядунов Дмитрий Александрович, Дюбуа Мартин
Формула / Реферат
1. Способ идентификации генотипа и подтипа вируса гепатита C на основе анализа области NS5B генома ВГС, включающий:
(а) обратную транскрипцию, совмещенную с ПЦР (ОТ-ПЦР), с использованием вирусной РНК в качестве матрицы и первой пары праймеров, специфичных к фрагменту области NS5B;
(б) асимметричную амплификацию фрагмента области NS5B с использованием в качестве матрицы продукта ОТ-ПЦР, полученного на стадии (а), второй пары специфичных праймеров и смеси четырех дезоксинуклеозидтрифосфатов, в которой один из четырех дезоксинуклеозидтрифосфатов является флуоресцентно меченым, в качестве субстрата с получением преимущественно одноцепочечного флуоресцентно меченого фрагмента;
(в) обеспечение биочипа для идентификации генотипа и подтипа ВГС, представляющего собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (б), и выбранную из группы, включающей а) генотип-специфичные для каждого из генотипов ВГС последовательности фрагмента области NS5B и б) подтип-специфичные для каждого из подтипов ВГС последовательности фрагмента области NS5B;
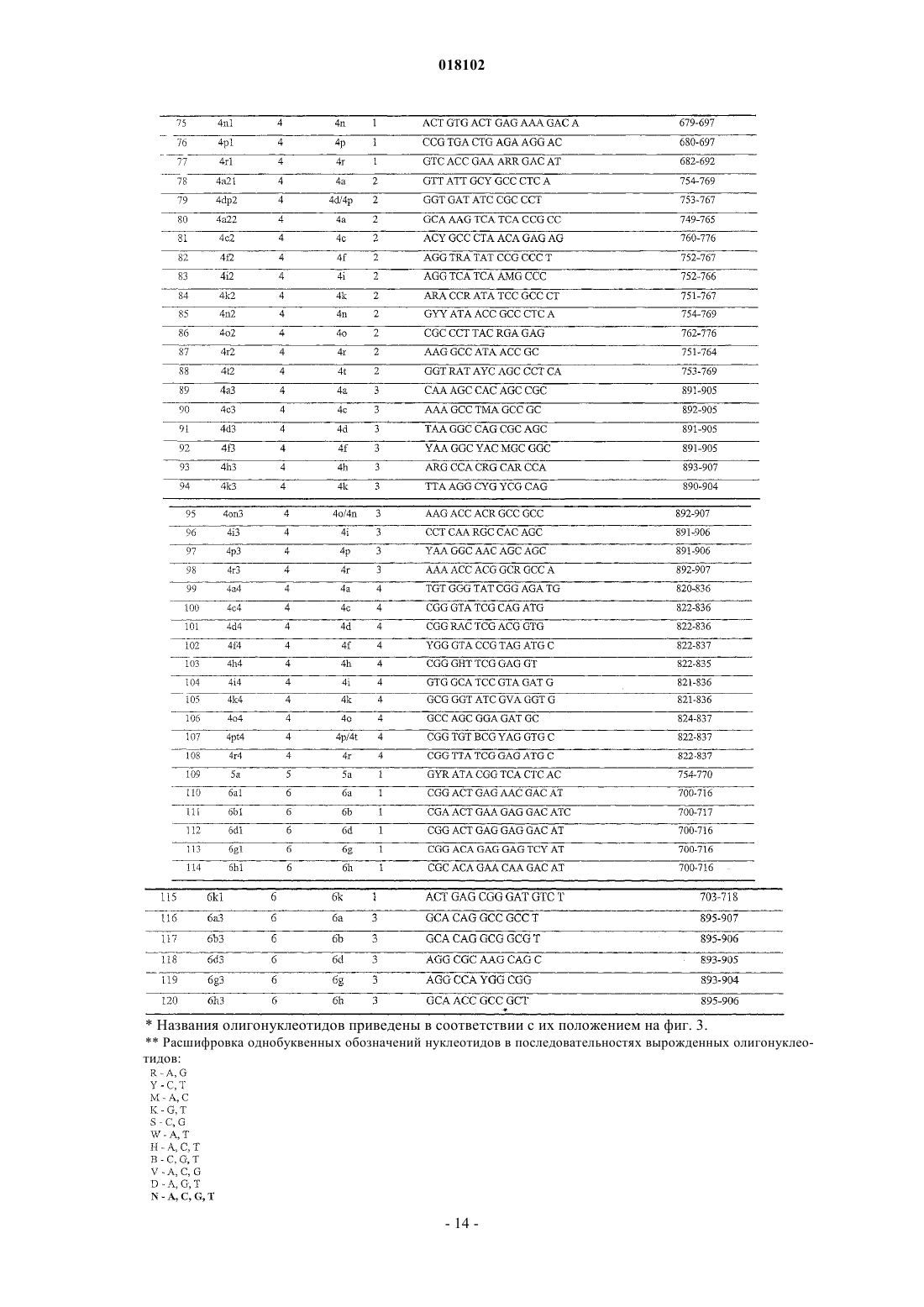
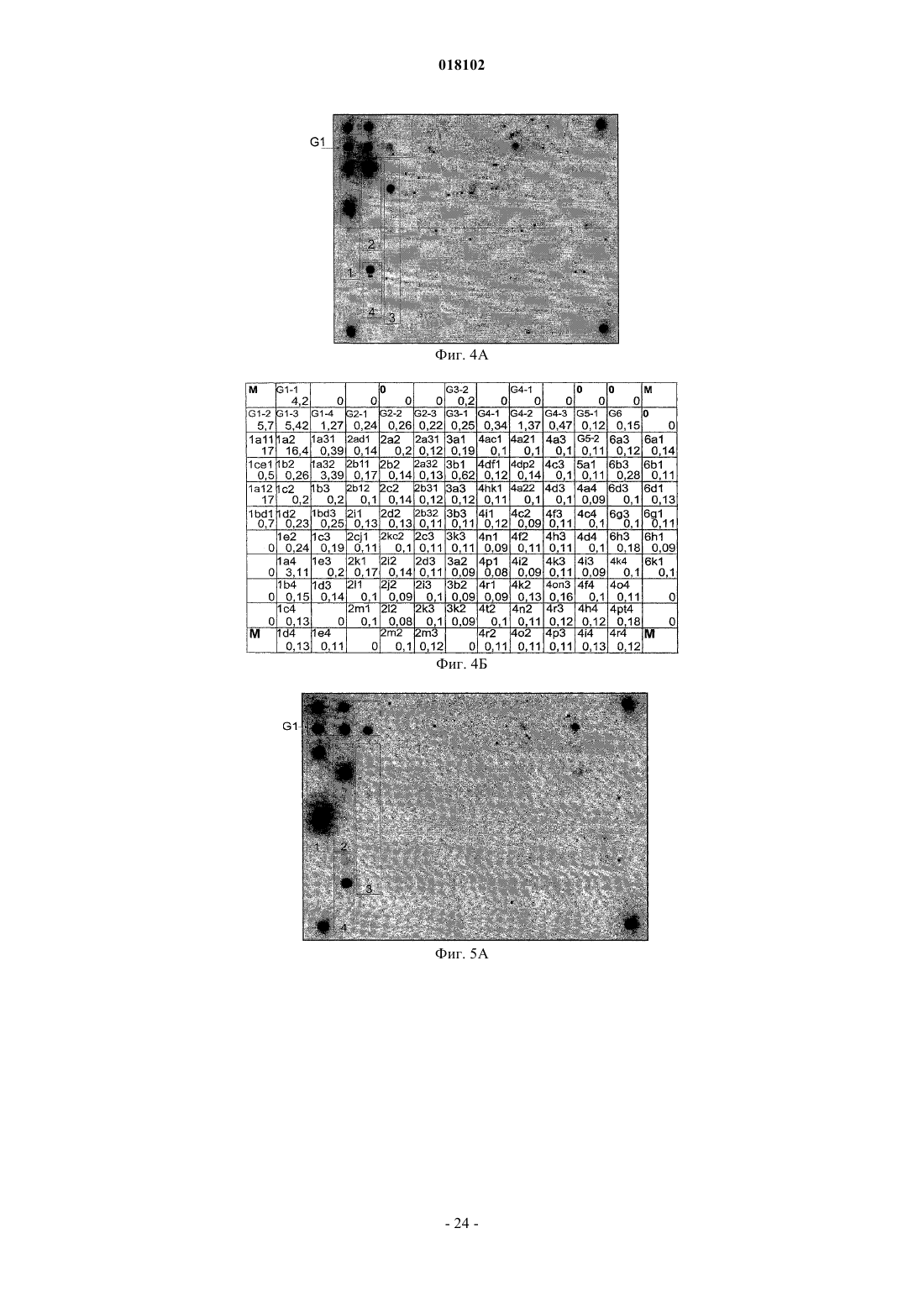
(г) гибридизацию амплифицированного меченого продукта, полученного на стадии (б), на биочипе с образованием дуплексов с иммобилизованными зондами в условиях, обеспечивающих разрешение в один нуклеотид между образующимися в результате гибридизации совершенными и несовершенными дуплексами;
(д) регистрацию и интерпретацию результатов гибридизации.
2. Способ по п.1, характеризующийся тем, что на стадии (а) используют первую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 122.
3. Способ по п.1, характеризующийся тем, что на стадии (б) используют вторую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 123.
4. Способ по п.1, характеризующийся тем, что на стадии (б) один из праймеров второй пары используют по меньшей мере в десятикратном молярном избытке по отношению ко второму праймеру.
5. Способ по п.1, характеризующийся тем, что на стадии (б) в качестве флуоресцентно меченого дезоксинуклеозидтрифосфата используют флуоресцентно меченый дезоксиуридинтрифосфат.
6. Способ по п.1, характеризующийся тем, что биочип представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации.
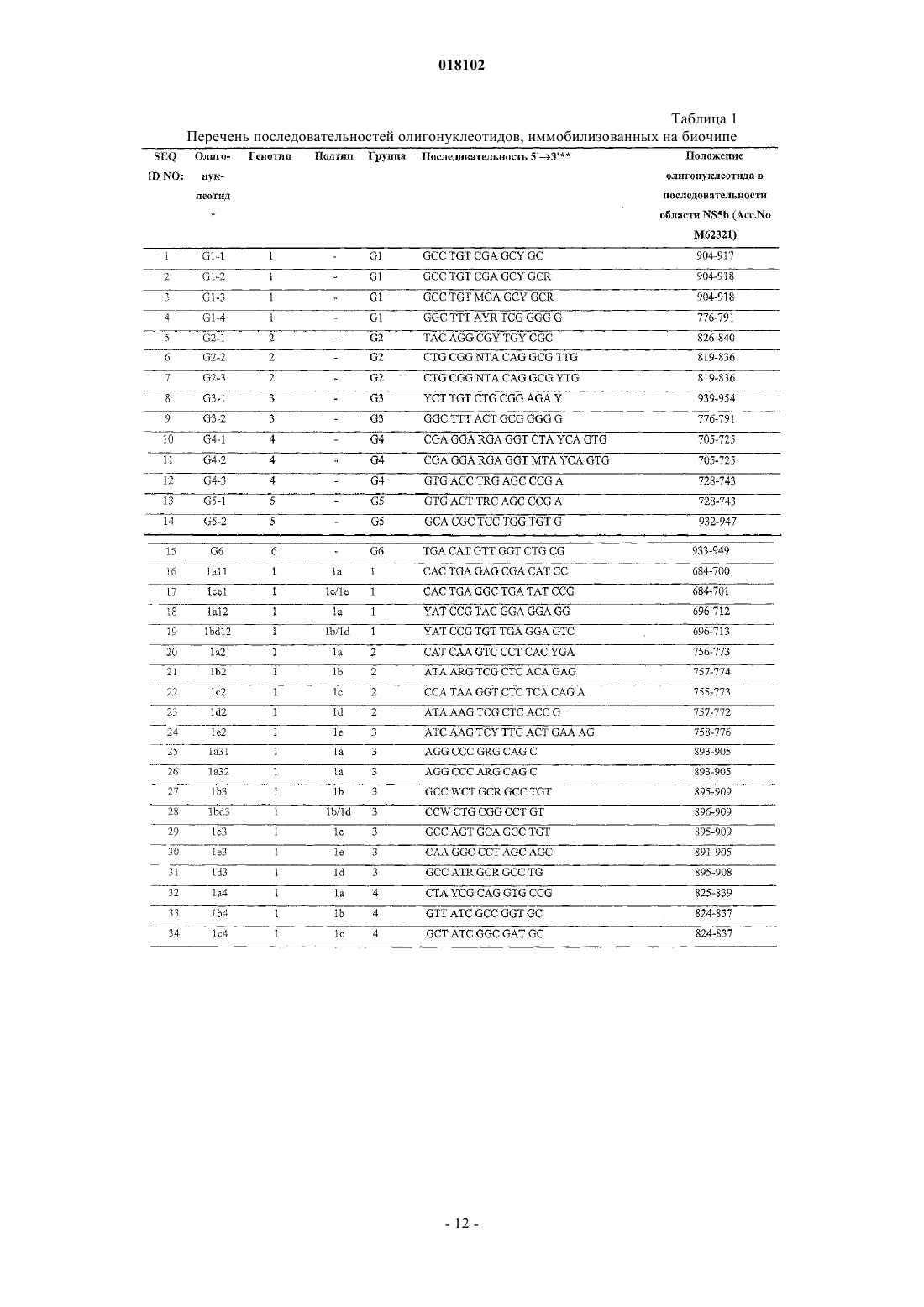
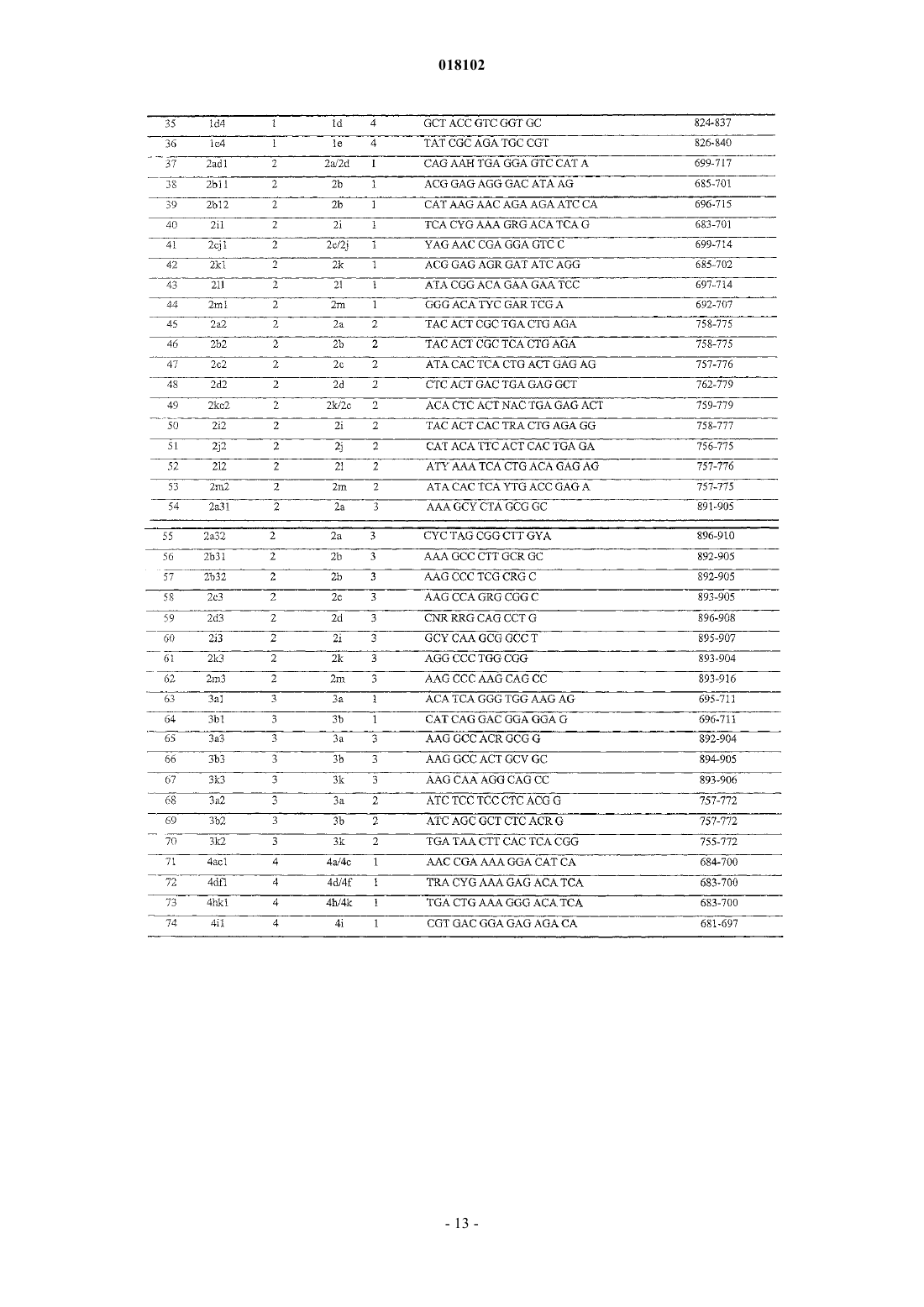
7. Способ по п.1, характеризующийся тем, что биочип содержит набор иммобилизованных олигонуклеотидов, последовательности которых представлены в SEQ ID NO: 1-120.
8. Способ по п.1, характеризующийся тем, что регистрацию результатов на стадии (д) проводят с помощью портативного анализатора флуоресценции и программного обеспечения, что позволяет использовать программную обработку интенсивностей сигналов с последующей интерпретацией результатов.
9. Способ по п.1, где интерпретацию зарегистрированных результатов на стадии (д) проводят в два этапа: на первом этапе анализируют сигналы в элементах биочипа, содержащих олигонуклеотидные зонды, специфичные к генотипам ВГС, тем самым идентифицируя генотип исследуемого образца; в случае идентификации генотипа на втором этапе анализируют только элементы биочипа, содержащие олигонуклеотидные зонды, специфичные к подтипам идентифицированного генотипа, независимо от наличия сигналов в элементах, содержащих зонды, специфичные к подтипам других генотипов.
10. Способ по п.1, дополнительно включающий оценку и прогнозирование тяжести протекания заболевания (острый/хронический цирроз, вероятность развития рака печени), определение терапевтической дозы лекарственных препаратов и длительности курса терапии и/или эпидемиологическое генотипирование на основе проведенной интерпретации результатов гибридизации.
11. Биочип для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, представляющий собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, причем последовательности зондов представлены в SEQ ID NO: 1-120.
12. Биочип по п.11, характеризующийся тем, что он представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации.
13. Набор олигонуклеотидных зондов для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, имеющих последовательности SEQ ID NO: 1-120.
14. Способ конструирования набора олигонуклеотидных зондов, используемых для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, предусматривающий раздельный выбор нескольких дискриминирующих зондов для каждого из генотипов и подтипов, последовательности которых комплементарны последовательностям различных участков исследуемого фрагмента области NS5B.
Текст
СПОСОБ ИДЕНТИФИКАЦИИ ГЕНОТИПА И ПОДТИПА ВИРУСА ГЕПАТИТА C НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ Изобретение относится к молекулярной биологии, вирусологии и медицине и обеспечивает способ идентификации генотипа и подтипа вируса гепатита C (ВГС) на основе анализа области NS5b генома ВГС с использованием дифференцирующего биочипа. Способ настоящего изобретения основан на двухстадийной ПЦР с получением флуоресцентно меченого преимущественно одноцепочечного фрагмента области NS5b с последующей гибридизацией этого фрагмента на биочипе, содержащем набор специфичных дискриминирующих олигонуклеотидов. Идентификацию генотипа и подтипа ВГС проводят путем определения специфичных последовательностей участков фрагмента области NS5b. Изобретение позволяет проводить анализ непосредственно из клинического образца, определять 6 генотипов и 36 подтипов ВГС, в том числе наиболее вирулентные и лекарственно устойчивые формы, снизить себестоимость анализа. Изобретение также касается биочипа, способа конструирования и набора олигонуклеотидных зондов, используемых при осуществлении способа.(FR), Заседателев Александр Сергеевич (RU), Изопет Жак (FR) Соболев А.Ю. (RU)(71)(73) Заявитель и патентовладелец: ФЕДЕРАЛЬНОЕ ГОСУДАРСТВЕННОЕ БЮДЖЕТНОЕ УЧРЕЖДЕНИЕ НАУКИ ИНСТИТУТ МОЛЕКУЛЯРНОЙ БИОЛОГИИ ИМ. В.А. ЭНГЕЛЬГАРДТА РОССИЙСКОЙ АКАДЕМИИ НАУК(ИМБ РАН) (RU); ЮНИВЕРСИТЕ ПОЛЬ САБАТЬЕ-ТУЛУЗ III; САНТР ОСПИТАЛЬЕ ЮНИВЕРСИТЕР ДЕ ТУЛУЗ (FR) Область техники, к которой относится изобретение Изобретение относится к области молекулярной биологии, вирусологии и медицины и касается способа идентификации генотипа и подтипа вируса гепатита C (ВГС) на основе анализа области NS5B генома ВГС с использованием дифференцирующего биочипа. Уровень техники ВГС относится к семейству РНК-содержащих вирусов Flaviviridae и вызывает у людей инфекционный процесс, наиболее часто встречаемым осложнением которого является гепатит, перерастающий в цирроз печени и гепатокарциному (Surveillance. Hepatitis. CDC Report61; Younossi Z., Kallman J.,Kincaid J. The effects of HCV infection and management on health-related quality of life. Hepatology. 2007Mar.; 45(3):806-16). Этим заболеванием поражено более чем 170 млн человек на Земле, и число инфицированных продолжает увеличиваться. По всему миру насчитывается порядка 1,5 млн случаев гепатокарциномы, вызванной инфекцией ВГС. Потери, связанные с этим заболеванием только в Соединенных Штатах, оцениваются в 200 млн долларов ежегодно. Распространнной современной тенденцией в лечении ВГС является применение комплексной терапии, включающей совместное введение мегадоз интерферона с коктейлем, содержащим как общие противовирусные препараты, так и один или два ингибитора размножения ВГС (ингибиторы специфической протеазы-геликазы и/или РНК-полимеразы) (Toniutto P., Fabris C., Bitetto D., Fornasiere E., Rapetti R.,Pirisi M. Valopicitabine dihydrochloride: a specific polymerase inhibitor of hepatitis C virus. Curr Opin Investighepatitis C virus NS3.4A protease control of antiviral immune defense. J. Biol. Chem. 2007 Apr.; 282(14):10792-803). Подобные коктейли повышают процент излечения, однако неминуемо ведут к селекции адаптивных мутантов ВГС, устойчивых к данным ингибиторам. Идентификация генотипа и подтипа исследуемого образца ВГС имеет важное значение для целей выявления лекарственной чувствительности, оценки длительности и эффективности противовирусной терапии, установления пути распространения вируса. Для определения генотипов и подтипов вируса гепатита C в настоящее время применяются следующие методы.I. Прямое определение нуклеотидной последовательности (секвенирование) 5'-некодирующей области генома вируса гепатита C с последующим анализом полученной последовательности путем сравнения с существующей базой данных, на основании которого делается заключение о принадлежности данного образца к определенному генотипу и подтипу (набор "TRUGENE HCV 5'-NC" (Bayer HealthCareV. Методы иммуноферментного анализа (Serotyping of hepatitis C virus):VI. Гетеродуплексный анализ с применением капиллярного электрофореза (heteroduplex mobilityVII. Метод инвазивных зондов (Invader Assay):VIII. Метод гнездовой ПЦР с последующим структурно-специфичным расщеплением (NestedIX. Высокоэффективная жидкостная хроматография (denaturing high-performance liquid chromatoXI. Метод ПЦР с детекцией в режиме реального времени с последующим анализом кривых плавления (Melting curve analysis): Doris M. Haverstick, Grant С. Bullock, and David E. Brans. 2004. Genotyping ofXII. Прямое определение нуклеотидной последовательности (секвенирование) области NS5B вируса гепатита C с последующим анализом полученной последовательности путем построения филогенетического дерева и определения генотипа и подтипа исследуемого образца на основании локализации анализируемой последовательности в одном из кластеров данного дерева (NS5B sequencing followed byApplication No. PCT/US2006/062582. Publication Date: 05.07.2007. Методы (I-IV, VI-XI) основаны на анализе генотип- и подтип-специфических последовательностей 5'-некодирующей области генома ВГС (5'-NC). Анализ последовательностей 5'-NC области дает возможность однозначно идентифицировать все 6 генотипов ВГС, однако обладает низкой эффективностью(менее 70%) в отношении дифференциации подтипов генотипа 1, в частности подтипа 1b, являющегося наиболее вирулентным и устойчивым к терапии рибавирином и интерфероном (K. Sandres-Saune, P.Sequences. J. Clin. Microbiol. Feb. 7). В настоящее время только анализ области NS5B позволяет идентифицировать подтип 1b со специфичностью, близкой к 100%. Более того, исследование последовательностей данной области дает возможность выявить значительно большее число подтипов, чем при анализе последовательностей 5'-NCFrance. J. Gen. Virol. Jan.; 86 (Pt. 1):107-14). Таким образом, проведение анализа последовательности области NS5B в настоящее время является необходимым для идентификации генотипа и подтипа исследуемого образца ВГС с целью выявления лекарственной чувствительности, оценки длительности и эффективности противовирусной терапии, ус-2 018102 тановления пути заражения (Laperche S., Saune K., Deny P., Duverlie G., Alain S., Chaix M.L., Gaudy C.,Lunel F., Pawlotsky J.M., Payan C., Pozzetto B., Tamalet C., Thibault V., Vallet S., Bouchardeau F., Izopet J.,Lefrere J.J. 2006. Unique NS5B hepatitis C virus gene sequence consensus database is essential for standardization of genotype determinations in multicenter epidemiological studies. J. Clin. Microbiol. Feb.; 44(2):614-6;Bioinformatics Feb. 1; 21(3):379-84). Метод секвенирования последовательности области NS5B ВГС с последующим филогенетическим анализом (XII) требует постановки реакций амплификации и секвенирования, проведения дополнительной очистки продуктов реакций после каждой из вышеперечисленных стадий и последующего анализа на автоматическом секвенаторе. Более того, последующий анализ хроматограмм, конструирование множественного выравнивания и построение филогенетических деревьев предъявляют повышенные требования к квалификации персонала, что препятствует широкому использованию данного подхода для анализа потока клинических образцов в условиях ординарной диагностической лаборатории. Метод выявления серотипов с помощью вариантов иммуноферментного анализа (V) позволяет выявлять лишь ограниченный набор генотипов и подтипов (1 а, 1b, 2 а, 2b, 3 а, и 4 а) и требует наличия высокоочищенных моноклональных антител для каждого серотипа. Также для перечисленных выше методов идентификации генотипов и подтипов отмечены следующие недостатки. Коммерческий набор INNO-LiPA (II) и его применение в сочетании с транскрипционноопосредованной амплификацией (X) отличается высокой себестоимостью и идентифицирует ограниченное количество подтипов. Метод с использованием генотип-специфических праймеров в пробирке (III) требует постановки независимых реакций по количеству генотипов (т.е. не менее 6) для выявления одного только генотипа. ПЦР-гетеродуплексный анализ с применением капиллярного электрофореза (VI) и метод гнездовой ПЦР с последующим структурно-специфичным расщеплением (VIII) трудоемки, занимают большое количество времени и требуют типовых стандартов на каждый определяемый генотип и/или подтип. Метод инвазивных зондов (VII) идентифицирует только генотип и не позволяет сделать заключение о подтипе, что является серьезным ограничением для его применения в клинической практике, где необходимо выявление лекарственно-устойчивых разновидностей ВГС (по крайней мере, 1b и 4d) для оценки эффективности и длительности терапии. Методы масс-спектрометрии (IV) и ВЭЖХ (IX) требуют наличия дорогостоящего оборудования,дополнительных стадий подготовки образца для анализа и идентифицируют ограниченное число подтипов. ПЦР с детекцией в режиме реального времени (XI) выявляет наличие только самых распространенных генотипов и подтипов, и при этом имеет весьма высокую стоимость для рутинного анализа. Таким образом, в данной области существует острая потребность в разработке способа идентификации генотипа и подтипа вируса гепатита С, который бы выгодно отличался от известных из уровня техники решений простотой проведения анализа, высокими специфичностью и информативностью в отношении числа идентифицируемых генотипов и подтипов, а также невысокой стоимостью. Раскрытие изобретения В результате проведенных обширных научных исследований авторы настоящего изобретения обнаружили, что задача разработки способа идентификации генотипа и подтипа вируса гепатита C может быть успешно решена путем использования биологических чипов (микрочипов) для анализа областиNS5B генома ВГС. Способ идентификации генотипа и подтипа вируса гепатита C (ВГС) на основе анализа областиNS5B генома ВГС на биочипах выгодно отличается от известных из уровня техники методов возможностью выявления всех 6 генотипов (1-6) и 36 подтипов ВГС (1 а-1 е, 2 а, 2b, 2 с, 2d, 2i, 2j, 2k, 2l, 2m, 3 а, 3b,3k,4a, 4c, 4d, 4f, 4h, 4i, 4k, 4n, 4o, 4p, 4r, 4t, 5a, 6a, 6b, 6d, 6g, 6h, 6k) в клинических образцах со специфичностью, близкой к 100%, за счет анализа последовательности области NS5B, а также низкой себестоимостью, малым временем, необходимым для получения результата. Метод не требует дорогостоящего оборудования и высококвалифицированного персонала. Данные, полученные с помощью метода гибридизации на биочипах, могут быть использованы для оценки и прогнозирования тяжести протекания заболевания (острый/хронический цирроз, вероятность развития рака печени), определения терапевтической дозы лекарственных препаратов и длительности курса терапии, а также для эпидемиологического генотипирования. В своем первом аспекте данное изобретение обеспечивает способ идентификации генотипа и подтипа ВГС на основе анализа области NS5B генома ВГС с использованием олигонуклеотидного биочипа. Способ настоящего изобретения основан на двухстадийной ПЦР с получением флуоресцентно меченого преимущественно одноцепочечного фрагмента области NS5B с последующей гибридизацией этого фрагмента на биочипе, содержащем набор специфичных дискриминирующих олигонуклеотидов, комплементарных вариантам последовательностей области NS5B, специфичных в отношении генотипов и подтипов. Способ предусматривает следующие стадии:(а) обратную транскрипцию, совмещенную с ПЦР (ОТ-ПЦР), с использованием вирусной РНК в качестве матрицы и первой пары праймеров, специфичных к фрагменту области NS5B;(б) асимметричную амплификацию фрагмента области NS5B с использованием в качестве матрицы продукта ОТ-ПЦР, полученного на стадии (а), второй пары специфичных праймеров и смеси четырех дезоксинуклеозидтрифосфатов, в которой один из четырех дезоксинуклеозидтрифосфатов является флуоресцентно меченым, в качестве субстрата, с получением преимущественно одноцепочечного флуоресцентно меченого фрагмента;(в) обеспечение биочипа для идентификации генотипа и подтипа ВГС, представляющего собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (б), и выбранную из группы, включающей: а) соответствующие специфичные для каждого из генотипов ВГС (генотип-специфичные) последовательности фрагмента области NS5B и б) соответствующие специфичные для каждого из подтипов ВГС (подтипспецифичные) последовательности фрагмента области NS5B;(г) гибридизацию амплифицированного меченого продукта, полученного на стадии (б), на биочипе с образованием дуплексов с иммобилизованными зондами в условиях, обеспечивающих разрешение в один нуклеотид между образующимися в результате гибридизации совершенными и несовершенными дуплексами;(д) регистрацию и интерпретацию результатов гибридизации. В одном из своих воплощений способ характеризуется тем, что на стадии (а) используют первую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 122. В еще одном из воплощений способ характеризуется тем, что на стадии (б) используют вторую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 123. В своем следующем воплощении способ характеризуется тем, что на стадии (б) один из праймеров второй пары используют по меньшей мере в десятикратном молярном избытке по отношению ко второму праймеру. В своем следующем воплощении способ характеризуется тем, что на стадии (б) в качестве флуоресцентно меченого дезоксинуклеозидтрифосфата используют флуоресцентно меченый дезоксиуридинтрифосфат. В еще одном из воплощений способ характеризуется тем, что биочип представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации. В своем следующем воплощении способ характеризуется тем, что биочип содержит набор иммобилизованных олигонуклеотидов, последовательности которых представлены в SEQ ID NO: 1-120. В следующем воплощении способ характеризуется тем, что регистрацию результатов на стадии (д) проводят с помощью портативного анализатора флуоресценции и программного обеспечения, что позволяет использовать программную обработку интенсивностей сигналов с последующей интерпретацией результатов. В следующем воплощении способ характеризуется тем, что интерпретацию зарегистрированных результатов на стадии (д) проводят в два этапа: на первом этапе анализируют сигналы в элементах биочипа, содержащих олигонуклеотидные зонды, специфичные к генотипам ВГС, тем самым идентифицируя генотип исследуемого образца; в случае идентификации генотипа на втором этапе анализируют только элементы биочипа, содержащие олигонуклеотидные зонды, специфичные к подтипам идентифицированного генотипа, независимо от наличия сигналов в элементах, содержащих зонды, специфичные к подтипам других генотипов. Наконец, в еще одном своем воплощении способ дополнительно включает оценку и прогнозирование тяжести протекания заболевания (острый/хронический цирроз, вероятность развития рака печени),определение терапевтической дозы лекарственных препаратов и длительности курса терапии и/или эпидемиологическое генотипирование на основе проведенной интерпретации результатов гибридизации. В своем следующем аспекте данное изобретение относится к биочипу для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, представляющему собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, причем последовательности зондов представлены в SEQ ID NO: 1-120. В следующем воплощении данного аспекта настоящего изобретения биочип характеризуется тем,что он представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации. Следующим аспектом данного изобретения является набор олигонуклеотидных зондов для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, имеющих последовательности в SEQ ID NO: 1-120. Наконец, еще одним аспектом данного изобретения является способ конструирования набора олигонуклеотидных зондов, используемых для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, предусматривающий раздельный выбор нескольких дискриминирующих зондов для каждого из генотипов и подтипов, последовательности которых комплементарны последовательностям различных участков исследуемого фрагмента области NS5B. Другие аспекты настоящего изобретения будут ясны из прилагаемых фигур, подробного описания и формулы изобретения. Краткий перечень фигур Для более ясного понимания сущности заявленного изобретения, а также для демонстрации его характерных черт и преимуществ далее приводится подробное описание изобретения со ссылками на фигуры чертежей, на которых: фиг. 1 представляет принципиальную схему проведения анализа области NS5B ВГС с целью идентификации генотипа и подтипа ВГС на биологическом микрочипе; фиг. 2 представляет схему выбора олигонуклеотидов для идентификации генотипов и подтипов на основе анализа фрагмента области NS5B генома ВГС; фиг. 3 представляет схему размещения дискриминирующих олигонуклеотидов на биочипе; фиг. 4 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 1, подтип 1a; фиг. 4 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 1, подтип 1a; фиг. 5 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 1, подтип 1b; фиг. 5 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 1, подтип 1b; фиг. 6 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 1, подтип 1e; фиг. 6 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 1, подтип 1e; фиг. 7 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 2, подтип 2 а; фиг. 7 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 2, подтип 2 а; фиг. 8 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 2, подтип 2i; фиг. 8 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 2, подтип 2i; фиг. 9 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 3, подтип 3a; фиг. 9 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 3, подтип 3 а; фиг. 10 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 4, подтип 4 а; фиг. 10 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 4, подтип 4 а; фиг. 11 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 4, подтип 4d; фиг. 11 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 4, подтип 4d; фиг. 12 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 5, подтип 5 а; фиг. 12 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в результате анализа образца ВГС, имеющего генотип 5, подтип 5 а; фиг. 13 А представляет флуоресцентную картину гибридизации, полученную на биочипе в результате анализа образца ВГС, имеющего генотип 6; фиг. 13 Б представляет распределение нормированных сигналов ячеек биочипа, полученное в ре-5 018102 зультате анализа образца ВГС, имеющего генотип 6. Осуществление изобретения Задача настоящего изобретения состоит в создании способа идентификации генотипа и подтипа вируса гепатита C на основе анализа области NS5B с помощью биологического микрочипа. В заявленном способе предложено использование процедуры обратной транскрипции, совмещенной с полимеразной цепной реакцией (ОТ-ПЦР), для амплификации последовательности фрагмента области NS5B; получение с наработанного продукта одноцепочечного флуоресцентно меченого фрагментаNS5B, при этом в качестве исходного образца может быть использована вирусная РНК, выделенная из клинического материала, такого как, например, кровь, плазма или биоптат печени. Заявленный способ также предусматривает использование оригинального олигонуклеотидного биочипа с иммобилизованными специфическими зондами, процедуры гибридизации, регистрации и интерпретации результатов. Принципиальная схема идентификации генотипа и подтипа исследуемого образца ВГС на биочипе. Схема анализа последовательности фрагмента области NS5B ВГС для идентификации генотипа и подтипа с помощью биочипа представлена на фиг. 1. Выделение РНК ВГС из клинического образца осуществляют с помощью известных в данной области способов (например, Hourfar M.K., Michelsen U., Schmidt М., Berger A., Seifried E., Roth W.K.Chem. 2005 Jul.; 51(7):1217-22) или любого специализированного коммерчески доступного набора реагентов для выделения РНК из крови, плазмы или биоптата печени, например QIAamp DSP Virus Kit(кат.60704, Qiagen, Германия), MagMAX AI/ND Viral RNA Isolation Kits (кат.AM1939, Ambion,США) или "Комплект реагентов для выделения РНК" (кат.05-013, ЗАО "НПФ ДНК-технология",Россия). Амплификацию фрагмента области NS5B генома ВГС на первом этапе выполняют с помощью реакции обратной транскрипции, совмещенной с ПЦР (ОТ-ПЦР). Для проведения ОТ-ПЦР могут быть использованы как различные системы, описанные, например, в Casabianca A., Orlandi С., Fraternale A.,Magnani M. A new one-step RT-PCR method for virus quantitation in murine AIDS. 2003 Journal ofRT-PCR Kit (кат.210210, Qiagen, Германия), Accuscript High-Fidelity RT-PCR Kit (кат.600180,Stratagene, США) и др. Праймеры для проведения первой стадии амплификации выбирают таким образом, чтобы они фланкировали наиболее полиморфный сегмент области NS5B, позволяющий дифференцировать существующие генотипы и подтипы ВГС. Предпочтительно, чтобы сегмент области NS5B, подлежащий амплификации, включал положения с 8256 по 8645 генома ВГС согласно Choo, Q.L., K.Н. Richman, J.H. Han, K.diversity of the hepatitis C virus. Proc. Natl. Acad. Sci. USA. 88: 2451-2455. Последовательности праймеров выбирают таким образом, чтобы проводить эффективную амплификацию РНК исследуемого сегмента области NS5B любого генотипа и, соответственно,подтипа ВГС. Для этого, например, может быть сконструировано множественное выравнивание всех доступных в базах данных нуклеотидных последовательностей области NS5B, находящихся по адресамhttp://hcv.lanl.gov/content/hcv-db. Затем находят наиболее консервативные для всех генотипов ВГС участки области NS5B и выбирают праймеры, специфичные к данным участкам. Используя специализированное программное обеспечение, например Oligo v. 6.3 (Molecular Biology Insights Inc., США) или Fast PCR(http://www.biocenter.helsinki.fi/bi/Programs/fastpcr.htm) или другие коммерчески доступные программы,или программы, свободно доступные в сети Internet, рассчитывают температуры плавления праймеров и,варьируя их длину, добиваются того, чтобы разброс температур отжига праймеров внутри пары не превышал 3-4 С. При подборе праймеров избегают таких последовательностей, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления. Каждый выбранный праймер должен обладать уникальной специфичностью в отношении анализируемого участка областиNS5B. Специфичность праймеров проверяется с помощью программного обеспечения, использующего поиск в базах нуклеотидных последовательностей по алгоритму BLAST (например,www.ncbi.nlm.nih.gov/BLAST). В частности, необходимо избегать таких последовательностей, которые способны продуктивно гибридизоваться (отжигаться) с последовательностями генома человека. На втором этапе получают преимущественно одноцепочечный флуоресцентно меченый продукт методом асимметричной ПЦР с использованием в качестве субстрата для построения цепи de novo смеси дезоксинуклеозидтрифосфатов, в которой один из дезоксинуклеозидтрифосфатов является флуоресцентно меченым. Как известно специалистам в данной области, смесь дезоксинуклеозидтрифосфатов для проведения ПЦР содержит dATP, dGTP, dCTP и dTTP, причем вместо dTTP может использоваться dUTP или смесь dTTP/dUTP в любой молярной пропорции. Любой из указанных дезоксинуклеозидтрифосфатов может нести флуоресцентную метку. Наиболее предпочтительно использование флуоресцентно меченого дезоксиуридинтрифосфата, что, с одной стороны, обусловливает эффективное включение данного субстрата во вновь синтезированную цепь ДНК в процессе ПЦР, с другой - обеспечивает возможность предотвращать получение ложных результатов за счет перекрестной кросс-контаминации ампликонами путем использования на первой стадии фермента урацил-ДНК-гликозилазы. Это имеет критическое значение при проведении массовых анализов в условиях специализированной лаборатории. В качестве флуоресцентного красителя может быть использован любой флуоресцентный краситель,который может быть химически включен в молекулу дезоксинуклеозидтрифосфата таким образом, чтобы не препятствовать в существенной степени прохождению полимеразной цепной реакции и последующей гибридизации полинуклеотидной молекулы, содержащей такие флуоресцентно меченые нуклеотидные остатки, с иммобилизованными олигонуклеотидными зондами. В случае флуоресцентно меченого дезоксиуридинтрифосфата, например, флуоресцентный краситель может быть присоединен к 5'-концу аминоаллильного производного dUTP. Примеры таких красителей хорошо известны специалисту в данной области техники и включают красители флуоресцеинового (TAMRA, ROX, JOE), родаминовогоFluorescence based strategies for genetic analysis. Chem. Commun., 2005, 5487-5502). Флуоресцентные красители коммерчески доступны, в частности от фирмы Molecular Probes, США. Наиболее предпочтительными являются красители, спектр возбуждения которых лежит в длинноволновой (красной) области спектра, что позволяет использовать для возбуждения флуоресценции недорогие источники возбуждающего излучения типа полупроводниковых лазеров. Флуоресцентно меченые дезоксинуклеозидтрифосфаты могут быть получены как в лабораторных условиях с использованием таких известных способов, как, например, Kuwahara M., Nagashima J., Hasegawa M., Tamura T., Kitagata R., Hanawa K., Hososhima S., Kasamatsu T., Ozaki H., Sawai H. Systematic(кат.PA55021, PA55032, PA55026, GE Healthcare, США). Праймеры для проведения второй стадии амплификации выбирают с учетом требований, изложенных выше, с тем отличием, что по меньшей мере один из праймеров выбирается внутри ПЦР-фрагмента,получаемого на первой стадии, что повышает специфичность реакции. Таким образом, получаемый продукт в конечном счете будет являться продуктом полугнездовой или гнездовой реакции амплификации. Размер амплифицируемого на второй стадии фрагмента специально не ограничивается до тех пор, пока это обеспечивает эффективную гибридизацию фрагмента с иммобилизованными на биочипе зондами. В том случае, когда биочип представляет собой биочип на основе гидрогелевых элементов, праймеры для проведения второй стадии амплификации выбирают так, чтобы размер амплифицируемого фрагмента составлял 100-800 п.о. Большая длина ПЦР-продукта, получаемого на второй стадии, затрудняет эффективную диффузию анализируемого фрагмента генома в гелевых элементах биочипа при гибридизации,что в конечном итоге может привести к уменьшению количества образовавшихся гибридизационных дуплексов и, как следствие, падению флуоресцентного сигнала в ячейках. При подборе праймеров следует учитывать тот факт, что на выходе реакции получают преимущественно одноцепочечные флуоресцентно меченые продукты, которые должны быть комплементарны олигонуклеотидам, иммобилизованным на биочипе. Поэтому в каждой паре праймер, добавляемый в избытке ("ведущий праймер"), выбирается из той цепи, последовательность которой комплементарна последовательностям иммобилизованных на биочипе олигонуклеотидов. Т.е. в случае, если олигонуклеотиды для иммобилизации выбраны из смысловой цепи, для формирования гибридизационных дуплексов в ячейках биочипа необходима преимущественная амплификация антисмысловой цепи, тем самым "ведущий праймер" выбирается из цепи,комплементарной последовательности гена (антисмысловая цепь), и наоборот. Для получения преимущественно одноцепочечного флуоресцентно меченого продукта ведущий праймер добавляют в молярном избытке по отношению ко второму праймеру. Предпочтительно молярный избыток является по меньшей мере десятикратным. При выборе дискриминирующих олигонуклеотидов для иммобилизации на биочипе с учетом размера и сложности анализируемой последовательности и, в частности, наличия повторов и протяженных гомополимерных последовательностей определяют длину дискриминирующих олигонуклеотидов, обеспечивающую их специфичность в отношении анализируемой последовательности. Выбор дискриминирующих олигонуклеотидов для генотипов и подтипов осуществляют следующим образом. В сконструированном множественном выравнивании последовательностей области NS5B для каждого генотипа создается консенсусная последовательность данного региона, на основании которой выбираются уникальные зонды, позволяющие однозначно идентифицировать каждый генотип. При этом в силу высокой вариабельности генома ВГС для повышения надежности способа по возможности выбирают несколько дискриминирующих зондов для каждого из генотипов, комплементарных различным участкам исследуемого фрагмента области NS5B. Для каждого из подтипов также создают консенсусную последовательность, а затем выбирают участки фрагмента исследуемой области NS5B, позволяющие дифференцировать максимальное количество подтипов внутри одного генотипа. Количество таких участков должно быть достаточно для обеспечения надежной идентификации каждого из подтипов. На основании последовательностей выбранных участков конструируют зонды для идентификации подтипов, при этом последовательность одного зонда может соответствовать сразу двум и более подтипам в отдельном дифференцирующем участке исследуемого фрагмента области NS5B. Стратегия выбора зондов для иммобилизации на биочипе схематично представлена на фиг. 2. Используя программное обеспечение, например Oligo v. 6.3 (Molecular Biology Insights Inc., США),рассчитывают температуры плавления олигонуклеотидов и, варьируя их длину, добиваются того, чтобы разброс температур плавления олигонуклеотидов составлял не более 2-3 С. Избегают таких олигонуклеотидов, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления. Дискриминирующие олигонуклеотиды иммобилизуют на подложке биочипа. В качестве подходящих подложек для изготовления биочипа могут использоваться активированная, например аминированная, поверхность предметных стекол (Adessi С., Matron G., Ayala G., Turcatti G., Mermod J., Mayer P.,Kawashima E. Solid phase DNA amplification: characterisation of primer attachment and amplification mechanisms Nucleic Acids Research. 2000. Vol. 51. 28(20):E87), пластиковые носители (Nikiforov Т., Rendle R.,Goelet P., Rogers Y., Kotewicz M., Anderson S., Trainor G., Knapp Michael R. Genetic Bit Analysis: a solidphase method for typing single nucleotide polymorphisms. Nucleic Acids Research. 1994, 22(20):4167-75),подложки с полимерными макропористыми носителями, такими как акриламид (Timofeev E., Kochetkovagels. Nucleic Acids Res. 1996, 24(16):3142-8), сефароза (Margulies M., Egholm M., Altman W.E. et al.,Genome sequencing in microfabricated high-density picolitre reactors. Nature. 2005, 437(7057):376-80) и др. Способы иммобилизации олигонуклеотидов на подложках хорошо известны в данной области и включают в себя следующее: хемосорбцию олигонуклеотидов и ДНК, содержащих тиоловую группу, на металлах (Mirkin C.A.,Letsinger R.L., Mucic R.C. and Storhoff J.J. A DNA-based method for rationally assembling nanoparticles intomacroscopic materials. Nature. 1996 Aug. 15; 382(6592):607-9); ковалентное связывание модифицированных олигонуклеотидов с функциональными группами поверхности, основанное на реакциях образования амидной (Healey B.G., Matson R.S., Walt D.R. Fiberoptic(Ghosh S.S., Musso G.F. Covalent attachment of oligonucleotides to solid supports. Nucleic Acids Res. 1987,15(13):5353-72), карбимидоильной (Matson R.S., Rampal J., Pentoney S.L. Anderson P.D., Coassin P. Biopolymer synthesis on polypropylene supports: oligonucleotide arrays. Anal Biochem. 1995, Jan 1; 224(1):110-6) связей и т.д.; фотохимически, химически и электрохимически индуцируемую сополимеризацию олигонуклеотидов, несущих непредельную группу, с мономерами, составляющими основу формируемой твердой фазы(Vasiliskov A.V., Timofeev E.N., Surzhikov S.A., Drobyshev A.L., Shick V.V. and Mirzabekov A.D.,Fabrication of microarray of gel-immobilized compounds on a chip by copolymerization. Biotechniques, 1999,27, 592-606). В предпочтительном воплощении настоящего изобретения используют биочип на основе гидрогелевых элементов. Способы получения таких биочипов включают полимеризацию аминомодифицированных олигонуклеотидов с образованием ковалентной связи с мономерами гидрогеля при соответствующих условиях (рН, температура, состав полимеризационной смеси и др.) (Rubina A.Y.,Pan'kov S.V., Dementieva E.I. et al. Hydrogel drop microchips with immobilized DNA: properties and methodsfor large-scale production. Anal. Biochem. 2004, 325:92-106). Наиболее предпочтительным является использование биочипов, содержащих гелевые элементы, которые наносятся на матрицу в виде капель диаметром от 80 до 300 мкм с периодом 150-500 мкм, без использования специальных приспособлений,например кварцевых масок. В качестве матрицы может использоваться как стеклянная подложка (предметное или покровное стекло), так и более доступные материалы, такие как пластик. Для иммобилизации олигонуклеотидов в гелевых элементах биочипа используется их совместная полимеризация с основными компонентами геля. В результате этой одностадийной реакции иммобилизуемые молекулы необратимо, ковалентно присоединяются к тем или иным мономерам растущей полимерной цепи и равномерно распределяются во всем объеме геля с высоким выходом (порядка 50% для олигонуклеотидов)(Rubina A.Y., Pan'kov S.V., Dementieva E.I. et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem. 2004, 325:92-106). Концентрация иммобилизованных олигонуклеотидных зондов может быть оценена прокрашиванием гелевых элементов биочипа красителем с низкой специфичностью к нуклеотидной последовательности ДНК (А.Л. Михейкин,А.В. Чудинов, А.И. Ярощук, А.Ю. Рубина, С.В. Паньков, А.С. Крылов, А.С. Заседателев,А.Д. Мирзабеков. Краситель с низкой специфичностью к нуклеотидной последовательности ДНК: применение для оценки количества олигонуклеотидов, иммобилизованных в ячейках биологических микрочипов. Молекулярная биология. 2003, 37(6):1061-70). ПЦР-продукты, полученные на второй стадии амплификации, гибридизуют на биочипе с иммоби-8 018102 лизованными дифференцирующими олигонуклеотидами, комплементарными участкам консенсусных последовательностей генотипов и подтипов фрагмента области NS5B. Гибридизацию проводят в растворе, содержащем буферный компонент для поддержания рН, соль для создания ионной силы и хаотропный (дестабилизирующий водородные связи) агент, в герметичной гибридизационной камере при температуре, зависящей от температуры плавления иммобилизованных на микрочипе дискриминирующих олигонуклеотидов. В качестве дестабилизирующего водородные связи агента могут быть использованы,например, гуанидин тиоцианат, мочевина или формамид. Выбор оптимальной температуры гибридизации проводят с учетом удобства практического применения системы. Дискриминирующие олигонуклеотиды, заявленные в настоящем изобретении, имеют температуру плавления в интервале от 42 до 44 С,что позволяет проводить гибридизацию при 37 С с использованием хаотропного агента. Температура 37 С удобна тем, что большинство клинических лабораторий оснащены термостатами, поддерживающими эту температуру. Изучаемый фрагмент ДНК образует совершенные гибридизационные дуплексы только с соответствующими (полностью комплементарными) олигонуклеотидами. Со всеми остальными олигонуклеотидами изучаемый фрагмент ДНК дает несовершенный дуплекс. Дискриминацию совершенных и несовершенных дуплексов выполняют путем сравнения интенсивностей флуоресценции ячеек, в которых образовались дуплексы. Интенсивность сигнала в ячейке, в которой образовался совершенный гибридизационный дуплекс (I сов), выше, чем в такой, где образовался несовершенный дуплекс (I несов). Проведение гибридизации при оптимальных условиях (температура, подобранная концентрация хаотропного агента и ионная сила гибридизационного буфера) позволяет добиться соотношения I сов/I несов 1,5 между двумя ячейками, содержащими зонды, принадлежащие одной группе и различающиеся на один нуклеотид. Регистрация результатов гибридизации на биочипах может быть осуществлена с помощью коммерческих сканирующих устройств - анализаторов флуоресценции биочипов, например GenePix 4000B(Axon Instruments, США), оснащенных соответствующим программным обеспечением для вычисления интенсивности флуоресцентных сигналов дискретных элементов биочипа и их последующей нормировки на фоновое значение, например "GenePix Pro", "Acuity" (Axon Instruments, США). Интерпретация результатов гибридизации может быть осуществлена визуально соотнесением фиксированной флуоресцентной картины биочипа и/или полученного распределения интенсивностей сигналов элементов биочипа к расположению специфичных дискриминирующих зондов в элементах биочипа(см. фиг. 3). Обладая распределением интенсивности сигналов в ячейках биочипа, выделяют максимальный сигнал среди элементов, содержащих генотип-специфичные олигонуклеотиды. Идентификация генотипа может быть осуществлена путем установления элемента биочипа, обладающего наибольшей интенсивностью флуоресценции, среди ячеек, содержащих зонды для определения генотипа ВГС. Идентификация подтипа исследуемого образца ВГС может быть осуществлена путем определения наибольших сигналов в ячейках, содержащих подтип-специфичные олигонуклеотиды, соответствующие установленному генотипу, в случае, если максимальные сигналы зарегистрированы по меньшей мере в двух различных ячейках, содержащих уникальные олигонуклеотиды, специфичные в отношении отдельного подтипа. Предпочтительно интерпретацию результатов гибридизации на биочипах осуществляют следующим образом. На первом этапе проводят выделение достоверных сигналов, т.е. сигналов в ячейках, где возможно образование совершенных дуплексов. Для этого нормированные сигналы интенсивности флуоресценции всех ячеек биочипа сортируют по возрастанию и сравнивают с усредненным сигналом(Iref) в ячейках, не содержащих олигонуклеотидов. Достоверными сигналами считаются такие, которые превосходят Iref по меньшей мере в 1,5 раза. Второй этап начинают с анализа отфильтрованных достоверных сигналов Gi в группах ячеек, содержащих олигонуклеотиды, специфичные к различным генотипам (i - номер генотипа). Выделяют максимальный сигнал внутри каждой группы ячеек Gimax и сравнивают между собой. Если сигнал Gimax в одной группе превосходит максимальные сигналы в остальных группах более чем в 1,5 раза (пороговое значение), то делается заключение о принадлежности исследуемого образца к данному генотипу. В случае если отношение сигналов среди Gimax не превышает порогового значения, то делается заключение о невозможности однозначной идентификации генотипа и о возможном присутствии в анализируемом образце смеси двух и более вариантов ВГС с различными генотипами. В случае если сигналы в группах, содержащих генотип-специфичные олигонуклеотиды, не проходят первичную фильтрацию относительно Iref, то делается заключение о низкой интенсивности сигналов и возможном отсутствии РНК ВГС в исследуемом образце. При этом идентификация подтипа не проводится. Таким образом, в случае если определен генотип на основании значения сигнала Gimax, то далее рассматриваются только группы ячеек, содержащих олигонуклеотиды, специфичные к подтипам, относящимся к идентифицированному генотипу. В соответствии с предложенной стратегий выбора зондов для идентификации подтипа олигонуклеотиды объединены в группы, соответствующие выбранным участкам исследуемого фрагмента NS5B, позволяющим дифференцировать максимальное число подтипов. При этом количество групп варьирует от 1 до 4 в зависимости от степени гомологии консенсусных последовательностей фрагмента NS5B различных подтипов и диктуется необходимостью надежной дифферен-9 018102 циации подтипов внутри генотипа. При идентификации подтипа сначала выбирают сигналы внутри каждой группы ячеек, превосходящие остальные сигналы в этой группе по меньшей мере в 1,5 раза, такой сигнал обозначается как Sixj (при этом i - номер генотипа, х - символьное обозначение подтипа в соответствии с принятой классификации подтипов ВГС, j - номер группы). В случае если в группе две и более ячейки имеют сигналы, различающиеся между собой менее чем в 1,5 раза, то выбирают все такие сигналы Sixj, Siyj и т.д. В результате получают набор ячеек из разных групп ix1, iy1, ix2, iz2, ix3 и т.д., сигналы в которых превосходят остальные сигналы в своих группах не менее чем в 1,5 раза. При этом если в полученном наборе присутствуют по меньшей мере 2 ячейки из разных групп, соответствующие одному подтипу, например ix1 и ix3 или ix1 и ixy2, то выдают заключение о принадлежности исследуемого образца к подтипу "x" генотипа "i". В случае если в полученном наборе ячейки из разных групп соответствуют разным генотипам, например ix1, iy2, iz3 или ix1, iyz3, тогда интенсивности сигналов данных ячеек сравнивают между собой. Если сигнал ячейки, соответствующей подтипу "х" одной группы превосходит сигналы ячеек других групп в 3 и более раза, то делают заключение о принадлежности исследуемого образца к подтипу "х". В случае, если отношение сигналов Six1/Siy2 не превышает трех, выдают заключение о том, что подтип не установлен и возможна принадлежность к подтипу "x" или подтипу "у". Такое же заключение выдается в случае, если максимальный сигнал в группе имеет ячейка, содержащая олигонуклеотид, специфичный к двум подтипам, например ixy1, при отсутствии достоверных сигналов в других группах ячеек. В случае если сигналы в группах, содержащих подтип-специфичные олигонуклеотиды, не проходят первичную фильтрацию относительно Iref, то считают, что подтип исследуемого образца не определен. Оценка длительности противовирусной терапии и прогноз течения заболевания могут быть сделаны на основании данных идентификации генотипа и подтипа. Так, в случае установления генотипа 1, приводящего к циррозу, хроническому гепатиту и гепатоцеллюлярной карциноме, длительность терапии пегилированным интерфероном и рибавирином должна составлять не менее 24 недель для подтипов 1a, 1c,1d, 1e, а в случае обнаружения подтипа 1b, устойчивого к интерферону - не менее 48 недель (Week K.Med. Virol. 2005 Sep.; 77(1):66-9). При этом в отличие от генотипа 1 для генотипа 4 характерно расщепление на значительно большее число подтипов (Nicot F., Legrand-Abravanel F., Sandres-Saune K., Boulestinstrains circulating in south-western France. J. Gen. Virol. Jan.; 86(Pt. 1):107-14). Клиническая значимость ряда из них, например 4 а и 4d, уже установлена: 4d является устойчивым к интерферону и требует пролонгированного курса лечения (не менее 48 недель) (Roulot D., Bourcier V., Grando V. et algenotype 4 infection. J. Viral. Hepat. 2007 Jul.; 14(7):460-7). Подтипы генотипов 2 и 3 ВГС являются чувствительными к терапии лекарственными препаратами и значительно реже приводят к хронизации процесса. Длительность курса терапии рибавирином и интерфероном для больных, инфицированных ВГС с данными генотипами, составляет от 6 до 12 недель. Помимо прогностических целей и оценки длительности терапии определение генотипа и подтипа дает информацию в плане этиологии инфекции. Субтипы 1 а, 3 а, 4 а, 4d чаще ассоциируются с "шприцевым" гепатитом у лиц, употребляющих парентеральные наркотики, в то время как генотип 2 и субтип 1b связан с гемотрансфузионным путем передачи (Simmonds P., Bukh J., Combet С. et al. Consensus proposalsfor a unified system of nomenclature of hepatitis C virus genotypes. Hepatology. 2005 Oct;42(4):962-73). Наконец, результаты, полученные с помощью способа настоящего изобретения, могут быть использованы для эпидемиологического генотипирования. Распределение генотипов и подтипов ВГС варьирует в различных географических регионах (Zein N.N. Clinical significance of hepatitis C virus genotypes. Clin.Microbiol. Rev. 2000, 13(2):223-35). Некоторые из подтипов являются универсальными, другие циркулируют лишь в пределах ограниченных географических зон. Подтип 1b превалирует на юге Европы, в Китае, Японии, России (50-80%). В США и странах Южной Америки, на севере Западной Европе чаще встречаются генотипы 1 а и 1b, за ним следуют генотипы 2 и 3. Генотип 4 превалирует в Северной и Центральной Африке, в то время как на юге континента основным является генотип 5. Генотип 3 встречается почти всюду и является основным в Австралии и Юго-Восточной Азии. Генотип 6 также распространен в Юго-Восточной Азии и является основным типом во Вьетнаме, одним из основных в Таиланде, Индонезии. Далее изобретение будет проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности заявленного изобретения, но не должны рассматриваться как ограничивающие данное изобретение. Примеры Пример 1. Биочип для идентификации генотипа и подтипа ВГС на основе анализа области NS5B. 1. Олигонуклеотиды для иммобилизации на биочипе и праймеры для амплификации были синтезированы на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США) и содержали спейсер со свободной аминогруппой 3'-Amino-Modifier С 7 CPG 500 (Glen Research, США) для последующей иммобилизации в гель. Изготовление биочипов производили по процедуре, описанной ранее(Rubina A.Y., Pan'kov S.V., Dementieva E.I. et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem. 2004, 325: 92-106). Биочипы содержали полусферические ячейки диаметром 100 мкм, отстоящие друг от друга на расстояние 300 мкм. Равномерность нанесения ячеек и их диаметр оценивали с помощью программного обеспечения "Тест-чип" (БиочипИМБ, Россия). Контроль качества микрочипов выполняли измерением концентраций иммобилизованных олигонуклеотидов. Биочипы окрашивали флуоресцентным красителем ImD-310 (Биочип-ИМБ), концентрацию иммобилизованных зондов оценивали, как было описано ранее (А.Л. Михейкин, А.В. Чудинов,А.И. Ярощук, А.Ю. Рубина, С.В. Паньков, А.С. Крылов, А.С. Заседателев, А.Д. Мирзабеков. Краситель с низкой специфичностью к нуклеотидной последовательности ДНК: применение для оценки количества олигонуклеотидов, иммобилизованных в ячейках биологических микрочипов. Молекулярная биология. 2003; 37(6): 1061-70). Структура биочипа. Биочип содержит 110 иммобилизованных олигонуклеотидов, список которых представлен в табл. 1,четыре маркерные точки (М) для правильного позиционирования (захвата изображения), выполняемого компьютерной программой, и 4 ячейки пустого геля (0), необходимые для вычисления референсного(фонового) значения интенсивности флуоресценции Iref. Расположение иммобилизованных на микрочипе олигонуклеотидов показано на фиг. 3. В двух верхних строках иммобилизованы олигонуклеотиды с индексом "G", позволяющие идентифицировать генотип ВГС. Биочип идентифицирует все шесть генотипов ВГС. Ниже иммобилизованы олигонуклеотиды, позволяющие идентифицировать подтипы внутри генотипа 1: 1a, 1b, 1c, 1d, 1e. Для надежной идентификации каждого из подтипов генотипа 1 сконструированы 4 группы олигонуклеотидов. Каждая группа соответствует отдельному участку внутри исследуемого фрагмента области NS5B; внутри генотипа 2: 2 а, 2b, 2 с, 2d, 2i, 2j, 2k, 2l, 2m. Для надежной идентификации каждого из подтипов генотипа 2 сконструированы 3 группы олигонуклеотидов. Каждая группа соответствует отдельному участку внутри исследуемого фрагмента области NS5B; внутри генотипа 3: 3 а, 3b, 3k. Для надежной идентификации каждого из подтипов генотипа 3 сконструированы 3 группы олигонуклеотидов. Каждая группа соответствует отдельному участку внутри исследуемого фрагмента области NS5B; внутри генотипа 4: 4 а, 4 с, 4d, 4f, 4h, 4i, 4k, 4n, 4o, 4p, 4r, 4t. Для надежной идентификации каждого из подтипов генотипа 4 сконструированы 4 группы олигонуклеотидов. Каждая группа соответствует отдельному участку внутри исследуемого фрагмента области NS5B; внутри генотипа 5: 5 а. Генотип 5 представляет собой единственный подтип 5 а, тем самым зонды,идентифицирующие генотип, выявляют и подтип 5 а; внутри генотипа 6: 6 а, 6b, 6d, 6g, 6h, 6k. Для надежной идентификации каждого из подтипов генотипа 6 сконструированы 2 группы олигонуклеотидов. Каждая группа соответствует отдельному участку внутри исследуемого фрагмента области NS5B. Названия олигонуклеотидов приведены в соответствии с их положением на фиг. 3.Расшифровка однобуквенных обозначений нуклеотидов в последовательностях вырожденных олигонуклеотидов:NS5B; получение одноцепочечных флуоресцентно меченых фрагментов методом асимметричной ПЦР. На первой стадии проводили обратную транскрипцию, совмещенную с амплификацией (ОТ-ПЦР) фрагмента области NS5B длиной 418 п.о. В 40 мкл смеси для ОТ-ПЦР вносили 10 мкл выделенного препарата РНК (конечный объем 50 мкл). Состав ПЦР-смеси: 1X ОТ-ПЦР-буфер: 70 мМ Трис-HCl, рН 8,3, 16,6 мМ (NH4)2SO4, 7,5 мМ MgCl2; 200 мкМ каждого dATP, dCTP, dGTP, dUTP (Силекс, Россия); смесь праймеров (последовательности представлены в табл. 2) Pr3f/ Pr2r в концентрации 200 нМ каждый; 10 ед. термостабильной ST-полимеразы (Силекс, Россия); 1 ед. урацил-ДНК-гликозилазы (Силекс, Россия); 10 ед. ингибитора РНКазы (Fermentas, Литва). Амплификацию проводили на термоциклере РТС-200 Dyad (MJ Research, США) со следующим режимом: обратная транскрипция при 50 С - 30 мин, затем 50 циклов ПЦР: 95 С - 30 с, 63 С - 30 с,72 С - 30 с; финальная элонгация при 72 С - 10 мин. 1 мкл реакционной смеси, полученной после первой стадии ПЦР, использовали в качестве матрицы при проведении второй стадии. Вторую стадию ПЦР проводили в полугнездовом варианте, с праймерами P3f/Pr5r, фланкирующими район области NS5B длиной 382 п.о. Последовательности праймеров приведены в табл. 2. Состав ПЦР-смеси (25 мкл): 1X ПЦР-буфер: 10 мМ KCl,10 мМ Трис-HCl (рН 8,3) (Силекс, Россия); 1,5 мМ MgCl2; 200 мкМ каждого dNTP (Силекс, Россия); 10 мкМ флуоресцентно меченого dUTP ("Биочип-ИМБ", Россия); смесь праймеров P3f/Pr5r в концентрации 20 нм/100 нМ соответственно; 10 ед. термостабильной Taq ДНК-полимеразы (Силекс, Россия). Амплификацию проводили на термоциклере РТС-200 Dyad (MJ Research, США) со следующим режимом: 95 С - 2 мин, затем 36 циклов: 95 С - 20 с, 60 С - 20 с, 72 С - 30 с, заключительная элонгация 72 С - 5 мин, 12 мкл полученного продукта использовали в гибридизации на биочипе. Таблица 2 Перечень последовательностей праймеров, используемых в двухстадийной ПЦР для амплификации фрагмента области NS5B Однобуквенное обозначение нуклеотида в последовательности вырожденного праймера: Y = C, T. Пример 3. Гибридизация амплифицированного меченого продукта на биочипе. К 12 мкл реакционной смеси, полученной после второй стадии ПЦР и содержавшей преимущественно одноцепочечные флуоресцентно меченые фрагменты ДНК, соответствующие исследуемому фрагменту области NS5B, добавляли концентрированный раствор гибридизационного буфера так, чтобы конечная концентрация гуанидина тиоцианата составляла 1 М, HEPES - 50 мМ, рН 7,5, ЭДТА - 5 мМ. Полученную смесь (32 мкл) помещали в гибридизационную камеру биочипа. Гибридизацию проводили при 37 С в течение 12-18 ч. По окончании гибридизации биочип трижды промывали дистиллированной водой при 37 С в течение 30 с и высушивали. Пример 4. Регистрация и интерпретация результатов гибридизации. Регистрация флуоресцентного изображения биочипа выполнялась на универсальном аппаратнопрограммном комплексе (УАПК) (ИМБ РАН, Россия) для анализа биочипов с использованием специализированного программного обеспечения "ImageWare" (Биочип-ИМБ, Россия). Интерпретацию результатов осуществляли согласно приведенному выше алгоритму, реализованному в модуле программного обеспечения для анализа флуоресцентных изображений биочипов Пример 5. Анализ области NS5B генома вируса гепатита С, имеющего подтип 1 а, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 4 А представлена гибридизационная картина биочипа. Фиг. 4 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,62. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G1, содержащих зонды, специфичные в отношении генотипа 1, превосходят Iref в 1,5 раза и более. Аналогичная ситуация с сигналом в ячейке G4-2 (1,37). Сигналы в других группах ячеек, содержащих генотип-специфичные зонды, были близки к фоновым. Далее, выбирается максимальный сигнал из группы G1, G1-2 (5,7). Сигнал в данной ячейке превосходит сигнал G4-1 более чем в 1,5 раза. Следовательно, последовательность РНК анализируемого образца относится к генотипу 1. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 1, выявляет следующее: в группе 1 максимальные (т.е. превосходящие пороговое значение 1,5 по отношению к другим ячейкам) сигналы имеют ячейки 1a11 (17,0) и 1 а 12 (17,0); в группе 2 - ячейка 1 а 2 (16,4); в группе 3 - ячейка 1 а 32 (3,4); в группе 4 - ячейка 1 а 4 (3,1). Таким образом, во всех группах максимальный сигнал имеют ячейки, содержащие зонды, специфичные в отношении подтипа 1 а. Следовательно, анализируемый образец относится к подтипу 1a. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 1 а. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 1 и подтип 1 а,что полностью совпадает с результатами секвенирования. Пример 6. Анализ области NS5B генома вируса гепатита С, имеющего подтип 1b, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадиипосле дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 5 А представлена гибридизационная картина биочипа. Фиг. 5 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,67. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G1, содержащих зонды, специфичные в отношении генотипа 1, превосходят Iref в 1,5 раза и более (максимальный сигнал имеет ячейка G1-3 (5,69. Сигналы в других группах ячеек, содержащих генотип-специфичные зонды, были близки к фоновым. Следовательно, последова- 16018102 тельность РНК анализируемого образца относится к генотипу 1. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 1, выявляет следующее: в группе 1 максимальный (т.е. превосходящий пороговое значение 1,5 по отношению к другим ячейкам) сигнал имеет ячейка 1bd1 (18,0); в группе 2 - ячейка 1b2 (16,3); в группе 4 - ячейка 1b4 (3,9); в группе 1 совершенный дуплекс с исследуемой ДНК формирует также олигонуклеотид, последовательность которого является универсальной для подтипов 1b и 1d. Однако максимальный сигнал зарегистрирован также в ячейках, содержащих зонды, специфичные только для подтипа 1b - 1b2 и 1b4. Следовательно, анализируемый образец относится к подтипу 1b. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 1b. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 1 и подтип 1b,что полностью совпадает с результатами секвенирования. Пример 7. Анализ области NS5B генома вируса гепатита С, имеющего подтип 1 е, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 6 А представлена гибридизационная картина биочипа. Фиг. 6 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,39. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G1, содержащих зонды, специфичные в отношении генотипа 1, превосходят Iref в 1,5 раза и более. Сигналы в других группах ячеек, содержащих генотип-специфичные зонды,близки к фоновым. Максимальный сигнал в группе G1 имеет ячейка G1-3 (4,82). Следовательно, последовательность РНК анализируемого образца относится к генотипу 1. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 1, выявляет следующее: в группе 1 максимальный (т.е. превосходящий пороговое значение 1,5 по отношению к другим ячейкам) сигнал имеет ячейка 1 се 1(18,0); в группе 2 - ячейка 1 е 2 (8,46); в группе 3 - ячейка 1 е 3 (3,52); в группе 4 - ячейка 1 е 4 (2,18). В группе 1 совершенные дуплексы с исследуемой ДНК формирует олигонуклеотид, последовательность которого является универсальной для подтипов 1 с и 1 е. Однако максимальный сигнал зарегистрирован также в ячейках, содержащих зонды, специфичные только для подтипа 1 е-1 е 2, 1 е 3 и 1 е 4. Следовательно, анализируемый образец относится к подтипу 1 е. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 1 е. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 1 и подтип 1e,что полностью совпадает с результатами секвенирования. Пример 8. Анализ области NS5B генома вируса гепатита С, имеющего подтип 2 а, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 7 А представлена гибридизационная картина биочипа. Фиг. 7 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,25. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G2, содержащих зонды, специфичные в отношении генотипа 2, превосходят Iref в 1,5 раза и более. Сигнал в ячейке G6 (2,01) также является достоверным относительно Iref. Максимальный сигнал в группе G2 имеет ячейка G2-2 (15,6), превосходящая сигнал в ячейке G6 более чем в 1,5 раза. Следовательно, последовательность РНК анализируемого образца относится к генотипу 2. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 2, выявляет следующее: в группе 1 максимальный (т.е. превосходящий пороговое значение 1,5 по отношению к другим ячейкам) сигнал имеет ячейка 2ad1 (15,2); в группе 2 - ячейка 2 а 2 (6,62); в группе 3 - ячейка 2 а 31 (2,33). Во всех трех группах максимальный сигнал имеют ячейки, содержащие зонды, специфичные в отношении подтипа 2 а. Следовательно, анализируемый образец относится к подтипу 2 а. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 2 а. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 2 и подтип 2 а,что полностью совпадает с результатами секвенирования. Пример 9. Анализ области NS5B генома вируса гепатита С, имеющего подтип 2i, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 8 А представлена гибридизационная картина биочипа. Фиг. 8 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,44. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что только сигналы в ячейках группы G2, содержащих зонды, специфичные в отношении генотипа 2,превосходят Iref в 1,5 раза и более. Максимальный сигнал в группе G2 имеет ячейка G2-3 (15,9). Следовательно, последовательность РНК анализируемого образца относится к генотипу 2. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 2, выявляет следующее: в группе 1 максимальный (т.е. превосходящий пороговое значение 1,5 по отношению к другим ячейкам) сигнал имеет ячейка 2i1 (2,3); в группе 2 - ячейка 2kc2 (10,9); в группе 3 - ячейка 2i3 (4,31); в группе 2 совершенные дуплексы с исследуемой ДНК формирует олигонуклеотид, последовательность которого является универсальной для подтипов 2 с и 2k. Однако в двух других группах максимальный сигнал принадлежит ячейкам, содержащим уникальные олигонуклеотиды, специфичные в отношении подтипа 2i. В соответствии с заявленным алгоритмом интерпретации анализируемый образец относится к подтипу 2i. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 2i. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 2 и подтип 2i,что полностью совпадает с результатами секвенирования. Пример 10. Анализ области NS5B генома вируса гепатита С, имеющего подтип 3 а, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 и.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 9 А представлена гибридизационная картина биочипа. Фиг. 9 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,52. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G3, содержащих зонды, специфичные в отношении генотипа 3, превосходят Iref в 1,5 раза и более. Сигнал в ячейке G4-2 (0,85) также превосходит Iref в 1,5 раза. Сигналы в других группах ячеек, содержащих генотип-специфичные зонды, близки к фоновым. Максимальный сигнал в группе G3 имеет ячейка G3-1 (15,4), сигнал в которой превосходит сигнал в G4-2 более чем в 1,5 раза. Следовательно, последовательность РНК анализируемого образца относится к генотипу 3. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 3, выявляет следующее: в группе 1 максимальный (т.е. превосходящий пороговое значение 1,5 по отношению к другим ячейкам) сигнал имеет ячейка 3 а 1 (16,2); в группе 2 - ячейка 3 а 2 (4,69); в группе 3 - ячейка 3 а 3 (1,74). Таким образом, во всех группах максимальный сигнал имеют ячейки, содержащие зонды, специфичные в отношении подтипа 3 а. Следовательно, анализируемый образец относится к подтипу 3 а. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 3 а. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 3 и подтип 3 а,что полностью совпадает с результатами секвенирования. Пример 11. Анализ области NS5B генома вируса гепатита С, имеющего подтип 4 а, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 10 А представлена гибридизационная картина биочипа. Фиг. 10 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вы- 19018102 числения усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,55. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что только сигналы в ячейках группы G4, содержащих зонды, специфичные в отношении генотипа 4,превосходят Iref в 1,5 раза и более. Сигналы в остальных ячейках, содержащих генотип-специфичные олигонуклеотиды, были близки к фоновым. Максимальный сигнал в группе G4 зарегистрирован в ячейкеG4-3 (17,9). Следовательно, последовательность РНК анализируемого образца относится к генотипу 4. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 4, выявляет следующее: в трех группах зондов, специфичных к генотипу 4, максимальные сигналы имеют ячейки, содержащие зонды для выявления подтипа 4 а: 4 ас 1 (16,3), 4 а 21 (1,08), 4 а 4 (14,1). В группе 3 максимальный сигнал имеет ячейка 43 (1,21), однако в соответствии с описанным алгоритмом анализируемый образец относится к подтипу 4 а. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 4 а. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 4 и подтип 4 а,что полностью совпадает с результатами секвенирования. Пример 12. Анализ области NS5B генома вируса гепатита С, имеющего подтип 4d, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 11 А представлена гибридизационная картина биочипа. Фиг. 11 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,35. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате было получено,что сигналы в ячейках группы G4, содержащих зонды, специфичные в отношении генотипа 4, превосходят Iref в 1,5 раза и более. Сигналы в остальных ячейках, содержащих генотип-специфичные олигонуклеотиды, были близки к фоновым. Максимальный сигнал в группе G4 имеет ячейка G4-1 (17,4). Следовательно, последовательность РНК анализируемого образца относится к генотипу 4. Анализ в группах ячеек, содержащих зонды, специфичные для подтипов генотипа 4, выявляет следующее: в группе 1 максимальный сигнал имеет ячейка 4df1 (11,6); в группе 2 - ячейка 4dp2 (3,16); в группе 3 - ячейка 4d3 (1,95); в группе 4 - ячейка 4d4 (7,35). Во всех группах максимальный сигнал имеют ячейки, содержащие зонды, специфичные к подтипу 4d. Следовательно, анализируемый образец относится к подтипу 4d. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 4d. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 4 и подтип 4d,что полностью совпадает с результатами секвенирования. Пример 13. Анализ области NS5B генома вируса гепатита С, имеющего подтип 5 а, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 12 А представлена гибридизационная картина биочипа. Фиг. 12 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,28. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате получаем, что только сигналы в ячейках группы G5, содержащих зонды, специфичные в отношении генотипа 5, превосходят Iref в 1,5 раза и более. Максимальный сигнал в группе G5 имеет ячейка G5-1 (6,58). Следовательно, последовательность РНК анализируемого образца относится к генотипу 5. Поскольку генотип 5 имеет всего один подтип (5 а) и сигнал в ячейке 5 а 2, специфичной к данному подтипу, является достоверным, делается заключение,что исследуемый образец имеет подтип 5 а. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность попадает в кластер последовательностей подтипа 5 а. Таким образом, установлено, что анализируемый образец РНК ВГС имеет генотип 5 и подтип 5 а,что полностью совпадает с результатами секвенирования. Пример 14. Анализ области NS5B генома вируса гепатита С, имеющего генотип 6, способом гибридизации на биочипе. Образец крови, полученный от пациента, был подвергнут обработке набором для выделения вирусной РНК Qiamp viral RNA (Qiagen, Германия) согласно протоколу фирмы-производителя. Выделенный препарат РНК был использован в ОТ-ПЦР, как описано в примере 2. По окончании ОТ-ПЦР наличие амплифицированного фрагмента NS5B заданной длины (418 п.о.) было протестировано электрофорезом в агарозном геле, после чего продукт ОТ-ПЦР был разделен на 2 части, одна из которых была обработана и проанализирована в соответствии с методикой, описанной в примерах 2-4 (вторая стадия ПЦР с последующей гибридизацией на биочипе, отмывкой, регистрацией и интерпретацией флуоресцентной картины биочипа). Вторую часть продукта первой стадии после дополнительной очистки использовали в реакции секвенирования с последующим разделением на автоматическом секвенаторе, получением и коррекцией хроматограммы, анализом обработанной последовательности фрагмента NS5B, конструированием множественного выравнивания и филогенетического дерева, на основании которых осуществляли установление генотипа и подтипа. На фиг. 13A представлена гибридизационная картина биочипа. Фиг. 13 Б демонстрирует распределение нормированных гибридизационных сигналов ячеек биочипа. В соответствии с предложенным алгоритмом интерпретации результатов анализ начинается с вычисления усредненного сигнала (Iref) в ячейках, не содержащих олигонуклеотидов. В данном случае значение Iref составило 0,72. В соответствии с этой величиной и пороговым значением, равным 1,5, осуществляют фильтрацию сигналов в генотип- и подтип-специфических ячейках. В результате получаем, что сигналы в ячейке группы G6 (18,6), содержащей зонд, специфичный в отношении генотипа 6, превосходят Iref в 1,5 раза и более. Сигнал в ячейке G3-1 (5,2) также следует считать достоверным. Ячейка G6 превосходит сигнал в ячейке G3-1 более чем в 1,5 раза, что означает принадлежность РНК анализируемого образца к генотипу 6. Из двух групп, содержащих зонды, специфичные к подтипам генотипа 6, ни в одной из ячеек сигнал не достигает порогового значения относительно Iref. Следовательно, в данном образце подтип ВГС является неустановленным и классифицируется как 6 х. Методом секвенирования с последующим филогенетическим анализом было показано, что исследуемая последовательность не попадает ни в один из кластеров подтипов генотипа 6 и формирует отдельную ветвь филогенетического дерева, т.е. является неклассифицированной. Таким образом, результаты, полученные обоими методами, подтвердили невозможность однозначного установления подтипа данного образца. Таким образом, представленное изобретение позволяет идентифицировать генотип и подтип вируса гепатита C на основе анализа области NS5B с использованием биологического микрочипа. Способ позволяет идентифицировать все 6 генотипов и 36 подтипов вируса гепатита С, в том числе наиболее вирулентные и лекарственно устойчивые формы. Способ данного изобретения выгодно отличается от существующих в настоящее время аналогов высокой специфичностью в отношении идентификации подтипов генотипа 1, в частности подтипа 1b, а также простотой выполнения и низкой себестоимостью. Данные,полученные с помощью способа гибридизации на биочипах настоящего изобретения, могут быть использованы для прогнозирования и оценки тяжести протекания заболевания (острый/хронический цирроз,- 21018102 вероятность развития рака печени), определения терапевтической дозы лекарственных препаратов и длительности курса терапии, а также для эпидемиологического генотипирования. Все патенты, публикации, научные статьи и другие документы и материалы, цитируемые или упоминаемые в данном документе, включены в настоящее описание путем отсылки в такой степени, как если бы каждый из этих документов был включен путем отсылки индивидуально или приведен здесь в его полном виде. Хотя предпочтительные воплощения настоящего изобретения и их преимущества были подробно описаны выше, специалист в данной области сможет внести различные изменения, дополнения, не выходя при этом за рамки данного изобретения, которые определяются прилагаемой ниже формулой изобретения. ФОРМУЛА ИЗОБРЕТЕНИЯ 1. Способ идентификации генотипа и подтипа вируса гепатита C на основе анализа области NS5B генома ВГС, включающий:(а) обратную транскрипцию, совмещенную с ПЦР (ОТ-ПЦР), с использованием вирусной РНК в качестве матрицы и первой пары праймеров, специфичных к фрагменту области NS5B;(б) асимметричную амплификацию фрагмента области NS5B с использованием в качестве матрицы продукта ОТ-ПЦР, полученного на стадии (а), второй пары специфичных праймеров и смеси четырех дезоксинуклеозидтрифосфатов, в которой один из четырех дезоксинуклеозидтрифосфатов является флуоресцентно меченым, в качестве субстрата с получением преимущественно одноцепочечного флуоресцентно меченого фрагмента;(в) обеспечение биочипа для идентификации генотипа и подтипа ВГС, представляющего собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (б), и выбранную из группы, включающей а) генотип-специфичные для каждого из генотипов ВГС последовательности фрагмента области NS5B и б) подтип-специфичные для каждого из подтипов ВГС последовательности фрагмента области NS5B;(г) гибридизацию амплифицированного меченого продукта, полученного на стадии (б), на биочипе с образованием дуплексов с иммобилизованными зондами в условиях, обеспечивающих разрешение в один нуклеотид между образующимися в результате гибридизации совершенными и несовершенными дуплексами;(д) регистрацию и интерпретацию результатов гибридизации. 2. Способ по п.1, характеризующийся тем, что на стадии (а) используют первую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 122. 3. Способ по п.1, характеризующийся тем, что на стадии (б) используют вторую пару специфичных праймеров, последовательности которых представлены в SEQ ID NO: 121 и 123. 4. Способ по п.1, характеризующийся тем, что на стадии (б) один из праймеров второй пары используют по меньшей мере в десятикратном молярном избытке по отношению ко второму праймеру. 5. Способ по п.1, характеризующийся тем, что на стадии (б) в качестве флуоресцентно меченого дезоксинуклеозидтрифосфата используют флуоресцентно меченый дезоксиуридинтрифосфат. 6. Способ по п.1, характеризующийся тем, что биочип представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации. 7. Способ по п.1, характеризующийся тем, что биочип содержит набор иммобилизованных олигонуклеотидов, последовательности которых представлены в SEQ ID NO: 1-120. 8. Способ по п.1, характеризующийся тем, что регистрацию результатов на стадии (д) проводят с помощью портативного анализатора флуоресценции и программного обеспечения, что позволяет использовать программную обработку интенсивностей сигналов с последующей интерпретацией результатов. 9. Способ по п.1, где интерпретацию зарегистрированных результатов на стадии (д) проводят в два этапа: на первом этапе анализируют сигналы в элементах биочипа, содержащих олигонуклеотидные зонды, специфичные к генотипам ВГС, тем самым идентифицируя генотип исследуемого образца; в случае идентификации генотипа на втором этапе анализируют только элементы биочипа, содержащие олигонуклеотидные зонды, специфичные к подтипам идентифицированного генотипа, независимо от наличия сигналов в элементах, содержащих зонды, специфичные к подтипам других генотипов. 10. Способ по п.1, дополнительно включающий оценку и прогнозирование тяжести протекания заболевания (острый/хронический цирроз, вероятность развития рака печени), определение терапевтической дозы лекарственных препаратов и длительности курса терапии и/или эпидемиологическое генотипирование на основе проведенной интерпретации результатов гибридизации. 11. Биочип для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, представляющий собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, причем последовательности зондов представлены в 12. Биочип по п.11, характеризующийся тем, что он представляет собой биочип на основе гидрогелевых ячеек, полученный способом химически- или фотоиндуцируемой сополимеризации. 13. Набор олигонуклеотидных зондов для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, имеющих последовательности SEQ ID NO: 1-120. 14. Способ конструирования набора олигонуклеотидных зондов, используемых для получения биочипа для идентификации генотипа и подтипа ВГС на основе анализа области NS5B, предусматривающий раздельный выбор нескольких дискриминирующих зондов для каждого из генотипов и подтипов, последовательности которых комплементарны последовательностям различных участков исследуемого фрагмента области NS5B.
МПК / Метки
МПК: C12N 15/11, C12Q 1/68
Метки: подтипа, генотипа, биологическом, вируса, микрочипе, способ, идентификации, гепатита
Код ссылки
<a href="https://eas.patents.su/30-18102-sposob-identifikacii-genotipa-i-podtipa-virusa-gepatita-c-na-biologicheskom-mikrochipe.html" rel="bookmark" title="База патентов Евразийского Союза">Способ идентификации генотипа и подтипа вируса гепатита c на биологическом микрочипе</a>
Предыдущий патент: Способы лечения кожных язв
Следующий патент: Опалубка для призматических колонн
Случайный патент: Способ предотвращения или уменьшения вызываемого температурным градиентом изгиба конструктивного элемента