Биомаркер для детекции высокогорной адаптации и высокогорного отека легких
Номер патента: 23630
Опубликовано: 30.06.2016
Авторы: Мукерджи Митали, Аггарвал Шилпи, Прашер Бхавана, Паша Мохаммед Абдул Кадар
Формула / Реферат
1. Биомаркер, пригодный для прогнозирования предрасположенности индивидуума к высокогорной адаптации и высокогорному отеку легких (НАРЕ), имеющий однонуклеотидный полиморфизм С/Т в положении 27 в SEQ ID NO: 1 и Т/С в положении 27 в SEQ ID NO: 2 гена EGLN1.
2. Биомаркер по п.1, имеющий SEQ ID NO: 2, где частота встречаемости "С"-аллеля rs480902 y людей, у которых развивается высокогорный отек легких, существенно (значение р<0,05) выше таковой у людей, проживающих на большой высоте.
3. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "С"-аллеля rs479200 y людей, проживающих на большой высоте, существенно (значение р<0,05) выше таковой у людей, проживающих на незначительной высоте.
4. Биомаркер по п.1, имеющий SEQ ID NO: 2, где частота встречаемости "СС"-генотипа rs480902 y людей, у которых развивается высокогорный отек легких, существенно (значение р<0,05) выше таковой у людей, проживающих на большой высоте.
5. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "Т"-аллеля rs479200 y людей, у которых развивается высокогорный отек легких, существенно (значение р<0,05) выше таковой у людей, проживающих на большой высоте.
6. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "ТТ"-генотипа rs479200 y людей, у которых развивается высокогорный отек легких, существенно (значение р<0,05) выше таковой у людей, проживающих на большой высоте.
7. Биомаркер по п.1, имеющий SEQ ID NO: 2, где частота встречаемости "Т"-аллеля rs480902 y людей, проживающих на большой высоте, существенно (значение р<0,05) выше таковой у людей, проживающих на незначительной высоте.
8. Праймер, пригодный для амплификации биомаркера по пп.1-7, имеющий последовательность, выбранную из группы, состоящей из:


которая является последовательностью SNaPshot-праймера.
9. Способ получения биомаркера по п.1, включающий:
a) выделение геномной ДНК из субъекта, являющегося человеком;
b) конструирование и синтез прямого и обратного олигонуклеотидных праймеров, имеющих SEQ ID NO: 8 и 9, для плюс-цепи интрона 1 гена EGLN1;
c) амплификацию плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью SEQ ID NO: 2, имеющей SNP ID rs480902;
d) конструирование и синтез прямого и обратного олигонуклеотидных праймеров, имеющих SEQ ID NO: 5 и 6, для минус-цепи интрона 1 гена EGLN1;
e) амплификацию минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью SEQ ID NO: 1, имеющей SNP ID rs479200;
f) определение SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO: 7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно.
10. Способ детекции высокогорной адаптации и предрасположенности индивидуума к высокогорному отеку легких, включающий стадии:
a) выделения геномной ДНК из субъекта, являющегося человеком;
b) конструирования и синтеза прямого и обратного олигонуклеотидных праймеров, имеющих SEQ ID NO: 8 и 9, для плюс-цепи интрона 1 гена EGLN1;
c) амплификации плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью SEQ ID NO: 2, имеющей SNP ID rs480902;
d) конструирования и синтеза прямого и обратного олигонуклеотидных праймеров, имеющих SEQ ID NO: 5 и 6, для минус-цепи интрона 1 гена EGLN1;
e) амплификации минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью SEQ ID NO: 1, имеющей SNP ID rs479200;
f) определения SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO: 7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно;
g) подсчета частот встречаемости ТТ-, ТС- и СС-генотипов в популяциях со стадии (f) для установления взаимосвязи данных генотипов с высокогорной адаптацией и высокогорным отеком легких;
h) прогнозирования и статистического анализа различий в распределении аллельных вариантов (Т, С) в популяции, где ТТ-генотип rs479200 и СС-генотип rs480902 в гене EGLN1 связаны с высоким риском высокогорного отека легких, а СС-генотип rs479200 и ТТ-генотип rs480902 в гене EGLN1 связаны с низким риском высокогорного отека легких.
11. Набор для детекции высокогорной адаптации и предрасположенности к высокогорному отеку легких у человека, содержащий:
1) праймеры, имеющие SEQ ID NO: 5, 6, 7, 8, 9, 10;
2) подходящие реагенты;
3) техническую документацию.
12. Применение биомаркеров для прогнозирования предрасположенности индивидуума к высокогорной адаптации и высокогорному отеку легких (НАРЕ), имеющих однонуклеотидный полиморфизм С/Т в положении 27 в SEQ ID NO: 1 и Т/С в положении 27 в SEQ ID NO: 2 гена EGLN1.
Текст
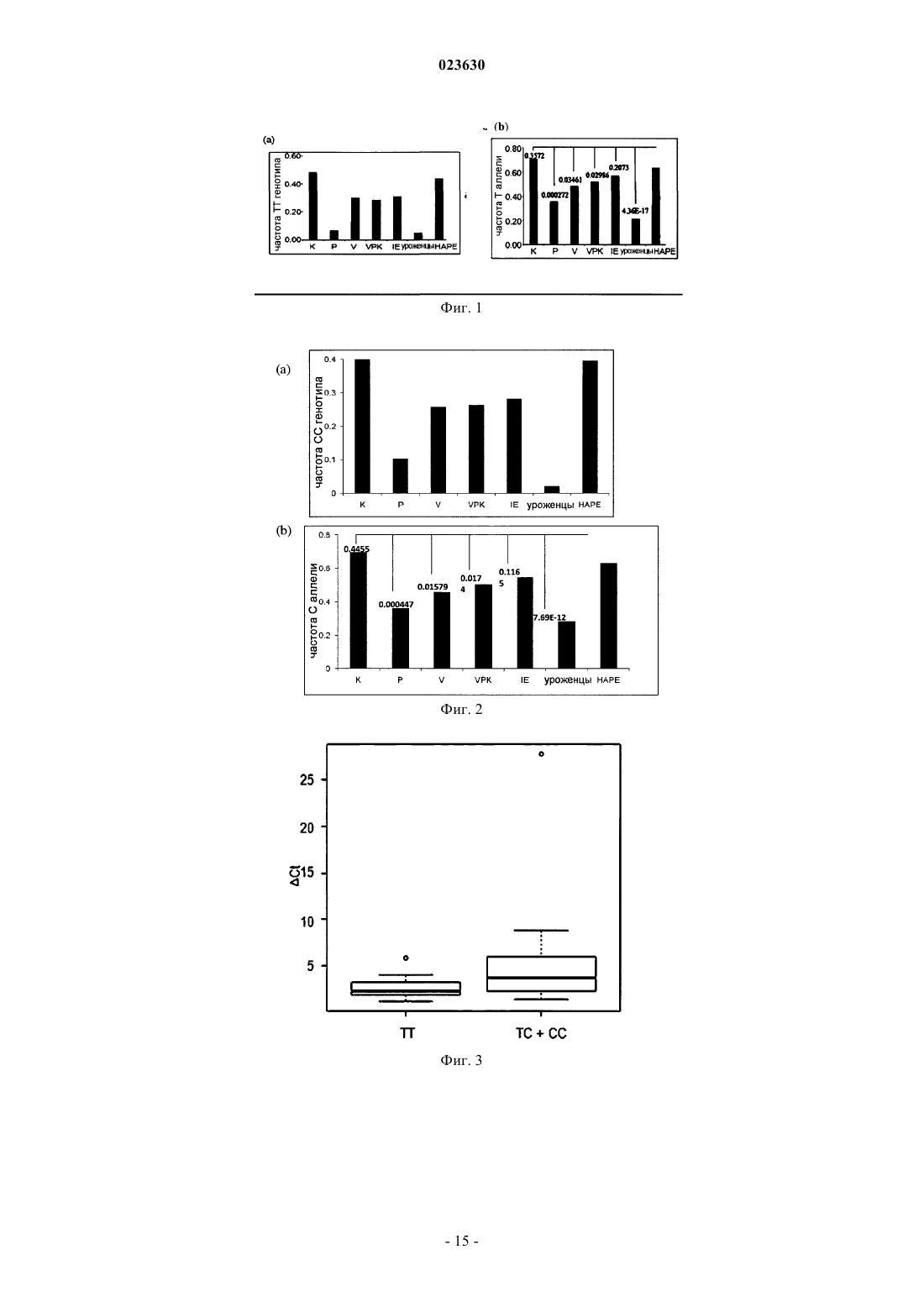
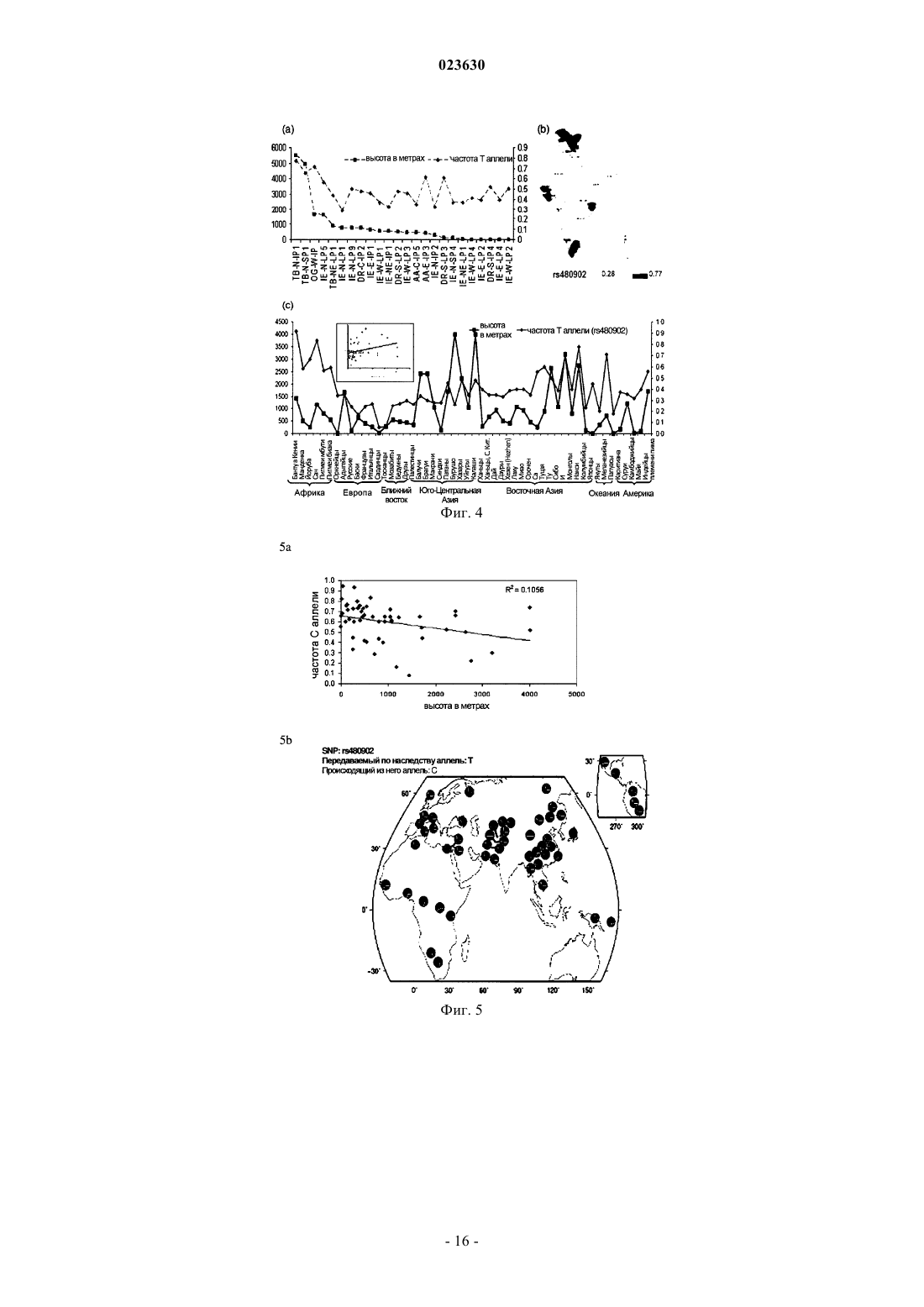
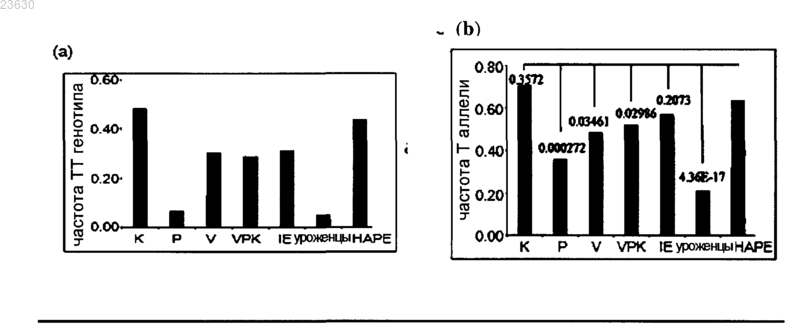
БИОМАРКЕР ДЛЯ ДЕТЕКЦИИ ВЫСОКОГОРНОЙ АДАПТАЦИИ И ВЫСОКОГОРНОГО ОТЕКА ЛЕГКИХ Настоящее изобретение относится к биомаркерам для детекции восприимчивости к высокогорной адаптации и гипоксии и способу их получения. Изобретение, в частности, относится к генным вариантам SNP ID rs479200 и rs480902 в первом интроне гена EGLN1 (пролилгидроксилазы 2) в качестве биомаркеров адаптации к большой высоте и предрасположенности к высокогорному отеку легких и восприимчивости к гипоксии, использование которых предусматривает новый комплексный подход с применением концепций фенотипирования согласно Аюрведе вместе с популяционной генетикой и геномикой заболеваний. Более конкретно, С-аллель SNP ID rs480902 и Т-аллель rs479200 гена EGLN1 чаще встречаются у пациентов с НАРЕ (высокогорным отеком легких) и практически отсутствуют у уроженцев горной местности. Согласно настоящему изобретению также предложены праймеры и способы, подходящие для детекции этих аллельных вариантов, с целью предсказания индивидуальной адаптируемости к большой высоте и гипоксии и/или с целью проведения генетического анализа гена EGLN1 в популяции. Область изобретения Настоящее изобретение относится к биомаркеру, полезному для предсказания предрасположенности индивида к высокогорной адаптации и высокогорному отеку легких (НАРЕ), характеризующемуся наличием однонуклеотидного полиморфизма С/Т в положении 27 в SEQ ID NO: 1 и Т/С в положении 27 в SEQ ID NO: 2 гена EGLN1. Изобретение также относится к способу детекции высокогорной адаптации и высокогорного отека легких с использованием маркеров, имеющих отношение к EGLN-1. Изобретение относится к биомаркерам, ассоциируемым с низким и высоким риском болезненного состояния, связанного с большой высотой, использование которых предусматривает новый комплексный подход с применением концепций фенотипирования согласно Аюрведе вместе с популяционной генетикой и геномикой заболеваний. Более конкретно, настоящее изобретение относится к аллельным вариантам гена пролилгидроксилазы 2 (PHD2) человека и разработке праймеров, подходящих для детекции этих аллельных вариантов с целью предсказания индивидуальной адаптируемости к большой высоте и гипоксии и/или с целью проведения генетического анализа гена EGLN1 в популяции. Предшествующий уровень техники При выявлении биомаркеров, связанных с восприимчивостью к заболеваниям и реакцией на внешнюю среду, сталкиваются со многими проблемами, обусловленными большой межиндивидуальной вариабельностью внутри популяций (Frazer и др., 2009; McClellan и King, 2010). Для описания популяций недостаточно использования характеристик этнической принадлежности ввиду существования большого разнообразия эндофенотипов в пределах популяций, которые имеют разную предрасположенность к различным заболеваниям и условиям окружающей среды. В сопоставимых по возрасту, полу и этнической принадлежности исследованиях методом случай-контроль наблюдается четкая зависимость от значительных различий в восприимчивости к заболеванию и/или адаптируемости между пациентами и контролями, если контроли не имеют очевидного клинического заболевания и в основном представляют собой гетерогенные эндофенотипы. Получается, что идентификация эндофенотипов среди нормальных контролен, соответствующих различной восприимчивости к заболеванию/адаптируемости, вероятно приведет к более эффективному обнаружению биомаркера. Аюрведа, древняя система индийской медицины, зафиксированная документально и практикуемая с 1500 г. до н.э., рассматривает межиндивидуальную вариабельность в рамках персонализированной и прогностической медицины. Эта система медицины фенотипически классифицирует индивидов на семь основных типов конституции, называемых пракрити, среди которых вата (V), питта (Р) и капха (K) являются самыми чистыми типами конституции,которые демонстрируют легко узнаваемые фенотипы (Sharma P.V. 2000; Prasher и др., 2008). Интеграция этого эндофенотипического подхода с генетическими исследованиями может "перекинуть мост" от фенотипа к генетическими вариациям. Поскольку наличие таких генетических вариаций также могло бы определять течение заболевания и приводить к развитию отдельных болезненных состояний, детекция их присутствия могла бы помочь в разработке персонализированного плана их лечения. Это также позволило бы осуществить разработку и распространение профилактического режима лечения, включающего в себя диету и образ жизни, чтобы избежать или отсрочить развитие таких болезненных состояний. В 2008 г. Prasher и др. продемонстрировали имеющееся различие в экспрессии всего генома и биохимических профилях для образцов аюрведических эндофенотипов среди этих чистых типов конституций. Среди по-разному экспрессирующегося набора генов было обнаружено значительное представительство генов хаба (активаторного хроматинового блока; англ. hub-узловая станция) и генов "домашнего хозяйства", что могло приводить к общесистемным эффектам. В более раннем исследовании (Prasher и др., 2008) было установлено, что ген EGLN1 присутствует среди 251 различным образом экспрессирующегося гена для трех чистых эндофенотипов (вата (V), питта (Р), капха (K среди нормальных индивидов индоевропейского (IE) происхождения.EGLN1 (пролилгидроксилаза 2) представляет собой "кислород-сенсорный" ген (oxygen sensor gen),который играет ключевую роль в гомеостазе кислорода путем регуляции HIF-1A (гипоксияиндуцибельного фактора) (Fong и Takeda, 2008) и ввиду этого имеет большое значение в целом ряде клеточных, физиологических и системных процессов. В нормоксических условиях EGLN1 гидроксилирует конститутивно экспрессирующийся HIF по двум его остаткам пролина, что приводит к его полиубиквитинированию под действием Е-3 лигазного комплекса фон Хиппеля-Линдау (VHL) и деградации под действием протеасомного комплекса (Semenza,2009). В условиях гипоксии EGLN1 неактивна, что приводит к стабилизации HIF, который индуцирует экспрессию генов, опосредующих адаптивные ответы на клеточном (посредством гликолитических ферментов, гемоксигеназы), местном (через сосудистый эндотелиальный фактор роста) и системном (через эритропоэтин) уровне (Smith и др., 2008). Вариации в EGLN1 могли бы способствовать различиям в физиологическом ответе на гипоксию, влияя тем самым на функциональные характеристики в условиях большой высоты. В настоящем изобретении рассматриваются вариации EGLN1, благодаря которым различают типы конституции у субъектов, у которых развивается НАРЕ, состояние, в норме присутствующее у не акклиматизировавшихся, временно находящихся на высотах свыше 2500 м людей, и которое является причи-1 023630 ной большинства случаев смерти от высотной болезни (Rodway и др., 2003). Данное заболевание характеризуется гипоксией, индуцирующей легочную вазоконстрикцию, вызванную эндотелиальной дисфункцией и задержкой интраваскулярной жидкости. Несмотря на то, что риску подвержены некоторые семейства и индивиды, те из них, предки которых длительное время проживали на большой высоте, подвержены меньшему риску. Кроме этого, индивиды, у которых был НАРЕ,подвержены большему риску повторных событий. Такие данные подтверждают наличие сильного генетического компонента восприимчивости к НАРЕ. Вполне вероятно, что длительное воздействие пребывания на большой высоте обеспечивает естественное положительное адаптивное давление на аллели,предупреждающее данную болезнь (Ahsan и др., 2004). Незамедлительный спуск с высоты пациентов с НАРЕ не только предупреждает обострение НАРЕ, но также улучшает патогенез данного заболевания(АСЕ (ангиотензин-превращающий фермент), eNOS (эндотелиальная синтаза оксида азота были изучены (Mortimer и др., 2004; Qadar Pasha и др., 2001; Morrell и др., 1999; Smith и др., 2008; Liu и др., 2007;Ahsan и др., 2004), ни в одном из исследований до сих пор не сообщалось об участии EGLN1 в НАРЕ. Эти исследования проводились как исследования связи методом случай-контроль. В недавнем полногеномном исследовании населения, проведенном среди жителей Тибета, сообщалось о том, что гаплотипы генов EGLN1 и PPARA (рецептор, активируемый пролифератором пероксисом, тип а) ассоциированы с НАА (Tatum и др., 2010). Однако там, в отличие от настоящего изобретения,не конкретизировано расположение SNP/область ДНК, авторами не проведено изучение ни НАРЕ, ни частоты этих гаплотипов в популяциях, проживающих на большой высоте в мировых масштабах. Ни в одном из исследований генетической предрасположенности в популяциях не рассматривались эндофенотипы, которые могли бы быть по-разному предрасположены к высокогорной адаптации (НАА). ОпределенияSNP - однонуклеотидные полиморфизмы (SNP) представляют собой наиболее общий тип генетической вариации. SNP представляет собой мутацию одной пары оснований в конкретном локусе, обычно состоящем из двух аллелей. Прогностический маркер - генетическая вариация (например, SNP), ассоциированная с риском или предотвращением конкретного заболевания либо ответом на терапию. Пракрита или тип конституции - пракрити или конституция тела индивида представляет собой проявление относительного соотношения трех энергий (трех дош) вата (V), питта (Р) и капха (К). Три доши работают совместно и поддерживают гомеостаз в течение всей жизни, начиная с оплодотворения. Каждой доше приписаны отдельные качества и функции. Вата - вата представляет собой одну из трех энергий, составляющих тридоши (Tridoshas). Она способствует проявлению формы, делению клеток, передаче сигналов, движению, выведению отходов, познанию, а также регулирует активности капха и питта. Капха - капха представляет собой одну из трех энергий, составляющих тридоши. Она отвечает за рост и поддержание структуры, сохранение и стабильность. Питта - питта представляет собой одну из трех энергий, составляющих тридоши. Она в первую очередь отвечает за метаболизм, терморегуляцию, гомеостаз энергии, пигментацию, зрение и общий контроль (host surveillance). Эндофенотипы - индивиды в пределах популяции, которые могут быть классифицированы на основании сходства физических, физиологических, поведенческих характеристик и т.д. Гипоксия - гипоксия представляет собой патологическое состояние, при котором организм в целом(генерализованная гипоксия) или участок тела (тканевая гипоксия) недостаточно снабжается кислородом.HGDP - проект "Разнообразие генома человека". СЕРН - Центр по изучению полиморфизма человека.IGVC - Индийский консорциум по геномным вариациям.IE-пул - пул индоевропейских больших популяций. Задача изобретения Основной задачей настоящего изобретения является разработка биомаркера, полезного для предсказания предрасположенности индивида к высокогорной адаптации и высокогорному отеку легких(НАРЕ), характеризующемуся наличием однонуклеотидного полиморфизма С/Т в положении 27 в SEQID NO: 1 (rs479200) и Т/С в положении 27 в SEQ ID NO: 2 (rs480902) гена EGLN1. Сущность изобретения Согласно настоящему изобретению предложены биомаркеры для детекции восприимчивости к высокогорной адаптации и гипоксии и способ их получения. В одном из воплощений настоящего изобретения предложены биомаркеры, представляющие собой варианты гена EGLN1 с однонуклеотидным полиморфизмом С/Т в положении 27 в SEQ ID NO: 1 предрасположенности индивида к высокогорной адаптации и высокогорному отеку легких (НАРЕ). В другом воплощении настоящего изобретения частота встречаемости "Т"-аллеля SNP ID rs479200 у пациентов с НАРЕ составляет 0,64, а у индивидов с пракрити капха составляет 0,71. В еще одном другом воплощении настоящего изобретения частота встречаемости "Т"-аллеляrs479200 y уроженцев горной местности составляет 0,21, а у индивидов с пракрити питта составляет 0,36. В еще одном другом воплощении настоящего изобретения частота встречаемости "Т"-аллеляrs479200 у пациентов с НАРЕ составляет 0,64, а у уроженцев горной местности составляет 0,21 (значение р составляет 4,3610-17). В еще одном другом воплощении настоящего изобретения частота встречаемости "Т"-аллеляrs479200 у пациентов с НАРЕ составляет 0,64, а у индивидов с пракрити питта составляет 0,36 (значение р составляет 0,000272). В следующем воплощении настоящего изобретения частота встречаемости "Т"-аллеля rs479200 у индивидов с пракрити капха составляет 0,71, а у индивидов с пракрити питта составляет 0,36 (значение р составляет 2,1710-4). В другом воплощении настоящего изобретения "ТТ"-генотип SNP ID rs479200 гена EGLN1 чаще встречается у пракрити капха и ассоциирован с высоким риском НАРЕ. В еще одном другом воплощении настоящего изобретения "ТТ"-генотип rs479200 был представлен преобладающим образом в типах пракрити капха, и также была обнаружена корреляция с высокой экспрессией гена EGLN1. В еще одном другом воплощении настоящего изобретения "ТС и СС"-генотип rs479200 был недостаточно представлен в типах пракрити капха, и также была обнаружена корреляция с низкой экспрессией гена EGLN1. В другом воплощении настоящего изобретения экспрессия гена EGLN1, ассоциированного с "ТС и СС"-генотипом rs479200, значительно отличалась от таковой для "ТТ"-генотипа (значение р составляет 0,017). В еще одном другом воплощении настоящего изобретения частота встречаемости "С"-аллеляrs480902 y пациентов с НАРЕ составляет 0,63, а у индивидов с капха составляет 0,69. В следующем другом воплощении настоящего изобретения частота встречаемости "С"-аллеля SNPID rs480902 у уроженцев горной местности составляет 0,28, а у индивидов с питта составляет 0,36. В другом воплощении настоящего изобретения частота встречаемости "С"-аллеля rs480902 y пациентов с НАРЕ составляет 0,63, а у индивидов с питта составляет 0,36 (значение р составляет 0,000447). В еще одном другом воплощении настоящего изобретения частота встречаемости "С"-аллеляrs480902 y пациентов с НАРЕ составляет 0,63, а у уроженцев горной местности составляет 0,28 (значение р составляет 7,6910-12). В другом воплощении настоящего изобретения частота встречаемости "С"-аллеля rs480902 y индивидов с капха составляет 0,69, а у индивидов с питта составляет 0,36 (значение р составляет 4,5510-4). В другом воплощении настоящего изобретения "СС"-генотип SNP ID rs480902 гена EGLN1 чаще встречается в типах пракрити капха и ассоциирован с высоким риском НАРЕ. В следующем воплощении настоящего изобретения "Т"-аллель SNP ID rs480902 и "С"-аллельrs479200 гена EGLN1 ассоциированы с низким риском НАРЕ и непосредственно зафиксированы у уроженцев горной местности. В следующем воплощении настоящего изобретения "С"-аллель SNP ID rs480902 и "Т"-аллельrs479200 гена EGLN1 чаще встречаются при НАРЕ и практически отсутствуют у уроженцев горной местности. В еще одном другом воплощении настоящего изобретения предложен праймер, полезный для амплификации биомаркеров, имеющий последовательности, выбранные из группы, состоящей из: В еще одном другом воплощении настоящего изобретения предложен способ получения биомаркеров, включающий:a) выделение геномной ДНК из субъекта, являющегося человеком;c) амплификацию плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью ID NO: 2,имеющей SNP ID rs480902;e) амплификацию минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью ID NO: 1,имеющей SNP ID rs479200;f) определение SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO:7 и 10 для SNP ID rs479200 и SNP ID rs480902, соответственно. В еще одном другом воплощении настоящего изобретения предложен способ детекции высокогорной адаптации и предрасположенности индивида к высокогорному отеку легких, включающий стадии:a) выделения геномной ДНК из субъекта, являющегося человеком;c) амплификации плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью ID NO: 2,имеющей SNP ID rs480902;e) амплификации минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью ID NO: 1,имеющей SNP ID rs479200;f) определения SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO:7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно;g) подсчета частот встречаемости ТТ-, ТС- и СС-генотипов в популяциях со стадии (f) для установления взаимосвязи данных генотипов с высокогорной адаптацией и высокогорным отеком легких;h) прогнозирования и статистического анализа различий в распределении аллельных вариантов (Т,С) в популяции, где ТТ-генотип rs479200 и СС-генотип rs480902 в гене EGLN1 связаны с высоким риском высокогорного отека легких, а СС-генотип rs479200 и ТТ-генотип rs480902 в гене EGLN1 связаны с низким риском высокогорного отека легких. В другом воплощении настоящего изобретения предложен набор для детекции высокогорной адаптации и предрасположенности индивида к высокогорному отеку легких, содержащий: 1 ) праймеры, имеющие SEQ ID NO: 5, 6, 7, 8, 9, 10; 2) подходящие реагенты; 3) техническую документацию. В другом воплощении настоящего изобретения предложено применение биомаркеров для предсказания предрасположенности индивида к высокогорной адаптации и высокогорному отеку легких (НАРЕ), характеризующейся наличием однонуклеотидного полиморфизма Т/С в положении 21782 в SEQ IDNO:3 (rs480902) и С/T в положении 12964 в SEQ ID NO: 4 (rs479200) гена EGLN1. В другом аспекте настоящего изобретения с помощью биомаркеров детектируют восприимчивость к высокогорной адаптации и гипоксии, и способ их применения включает: 1) эндофенотипирование нормальных здоровых людей для анализа типов пракрити и отбор участников исследования для геномного анализа; 2) выделение геномной ДНК и РНК с последующим генотипированием известными методами; 3) детекцию однонуклеотидных полиморфизмов в гене EGLN1 с использованием набора ПЦРпраймеров для SNP ID rs479200, имеющих SEQ ID NO: 5 и 6; 4) детекцию однонуклеотидных полиморфизмов в гене EGLN1 с использованием набора ПЦРпраймеров для SNP ID rs480902, имеющих SEQ ID NO: 8 и 9; 5) определение SNP y пациентов с НАРЕ и уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO: 7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно; 6) повторное секвенирование состоящего из 11746 п.о. участка интрона 1 гена EGLN1, начиная от положения на хромосоме chrl 229598510 до 229610256 (Assembly-Mar.2006 (NCBI36/hg18. Краткое описание графических материалов Фиг. 1 - связь между rs479200, имеющим отношение к высокогорной адаптации, и различными типами конституции, IE-пулом, уроженцами высокогорной местности и пациентами с НАРЕ:(a) Частота встречаемости "ТТ"-генотипа rs479200 y различных типов конституции (K, Р, V), VPK(V+P+K), IE, уроженцев высокогорной местности и пациентов с НАРЕ;(b) Частота встречаемости "Т"-аллеля rs479200 y различных типов конституции (K, Р, V), VPK, IE,уроженцев высокогорной местности и пациентов с НАРЕ. Числа над каждым из столбцов представляют собой значения р, полученные при сравнении каждой из групп с группой НАРЕ. Фиг. 2 - связь между rs480902, имеющим отношение к высокогорной адаптации, и различными типами конституции, IE-пулом, уроженцами высокогорной местности и пациентами с НАРЕ:(a) Частота встречаемости "СС"-генотипа rs480902 y различных типов конституции (K, Р, V), VPK,IE, уроженцев высокогорной местности и пациентов с НАРЕ;(b) Частота встречаемости "С"-аллеля rs480902 y различных типов конституции (K, Р, V), VPK, IE,уроженцев высокогорной местности и пациентов с НАРЕ. Числа над каждым из столбцов представляют собой значения р, полученные при сравнении каждой из групп с группой НАРЕ. Фиг. 3 - корреляция генотипов EGLN1, имеющих rs479200, с генной экспрессией. Ящичковая диаграмма, представляющая величины Ct генной экспрессии EGLN1 по данным ПЦР в режиме реального времени (RT PCR) в "ТТ"- и "ТС и СС"-генотипах rs479200 для аюрведических образцов. Фиг. 4 - распределение частоты встречаемости "Т"-аллеля rs480902 в различных IGVC- и HGDPпопуляциях, проживающих на разной высоте:b) Карта пространственной частоты встречаемости rs480902 в IGVC-популяциях. Градиент цвета,расположенный ниже карты, отображает диапазон наблюдаемых частот встречаемости "Т"-аллеля от минимума к максимуму.(с) Распределение частоты встречаемости в HGDP-панели из 52 популяций вместе с высотами их проживания. Разнообразные континентальные популяции, проживающие на большой высоте, избирательно сохраняют передаваемый по наследству "Т"-аллель. Фиг. 5 - распределение частоты встречаемости аллеля rs480902 в HGDP-популяциях.(а) Корреляция частоты встречаемости "Т"-аллеля rs480902 с увеличением высоты (R2 = 0,1056) в(b) Карта пространственной частоты встречаемости rs480902 в HGDP-популяциях, взятая из браузера для просмотра данных HGDP-отбора (HGDP selection browser). Частоты встречаемости передаваемого по наследству "Т"-аллеля, предпочтительно присутствующего в популяциях, проживающих на большой высоте, и происходящего из него "С"-аллеля представлены темными и светлыми оттенками, соответственно. Подробное описание изобретения Настоящее изобретение относится к детекции предрасположенности к НАРЕ и высокогорной адаптации. В частности, оно относится к аллельным вариантам гена EGLN1 (SEQ ID NO: 1 и SEQ ID NO: 2),который связан с гомеостазом кислорода посредством регуляции HIF-1A, гипоксия-индуцируемого фактора, и ввиду этого имеет важное значение в целом ряде клеточных, физиологических и системных процессов. Данные, изложенные в настоящем описании, демонстрируют, что "ТТ"-генотип rs479200 генаEGLN1 встречался чаще, и было обнаружено, что он ассоциирован с высоким риском НАРЕ (фиг. 1 а). Этот "ТТ"-генотип rs479200 был представлен преобладающим образом в типах пракрити капха, и также была обнаружена корреляция с высокой экспрессией гена EGLN1 (фиг. 3). Наряду с этим частота встречаемости "ТТ"-генотипа была значительно ниже для пракрити питта, и было обнаружено, что он практически отсутствует у индивидов - уроженцев горной местности. Таким образом, индивиды с пракрити питта являются носителями протективных "С"-аллелей, ответственных за адаптацию к большой высоте,в то время как тип пракрити капха является носителем "рисковых" "Т"-аллелей, ответственных за плохую адаптацию к большой высоте, соответственно (фиг. 1b). Для другого SNP rs480902 в гене EGLN1 было обнаружено, что "С"-аллель ассоциирован с высоким риском НАРЕ, в то время как "Т"-аллель ассоциирован с низким риском (защитой от) НАРЕ (фиг. 2b). Было обнаружено, что протективный "Т"аллель чаще встречается у индивидов с пракрити питта, а также у уроженцев горной местности, в то время как "рисковый" аллель чаще встречается в типах капха и у пациентов с НАРЕ. Важнейшим результатом данного изобретения является то, что разрозненные генетические линии, проживающие на большой высоте, совместно имеют один и тот же передаваемый по наследству протективный "Т"-аллельrs480902 в мировом масштабе (фиг. 4 и 5). Эндофенотипирование Опросник (рег.авторского права SW-2284/2005, дата регистрации 13 мая 2005 г.) для проведения клинического фенотипирования был разработан на основе аюрведической литературы по фенотипам и методам оценки пракрити (анализ основных конституций), подробное описание которых приведено в опубликованной ранее статье. Фенотипическая классификация, в целом, учитывает параметры, связанные с анатомическими особенностями, подобные телосложению, скелету, размеру и симметрии частей тела, физиологии, физической выносливости и способностям. Проводили скрининг 850 индивидов на предмет анализа конституции их тела (пракрити). Среди них были выявлены 96 индивидов с преобладающей в пракрити дошей, в том числе ватой (39), питтой(29) и капхой (28), и они были отобраны для сбора образцов. Эти индивиды из возрастной группы 18-40 лет (средний возраст 234 года) имели индоевропейское происхождение и включали приблизительно одинаковое число представителей обоих полов (Prasher и др., 2008). Генотипирование Исследуемые субъекты 1) 96 образцов от индивидов, классифицированных на основе типов их конституции, и в том числе индивидов с типами вата (39), питта (29) и капха (28). 2) 552 образца от 24 различных популяций, проживающих в Индии, из существующей панели Индийского консорциума по геномным вариациям (IGVC). Эти популяции охватывают различные этнические и языковые группы, проживающие в различных географических регионах, и представляют весь генетический спектр Индии. Они составляли часть от 55 популяций, которые были использованы в Фазе I Индийского проекта по геномным вариациям для установления генетического родства среди различных популяций в Индии (Индийский консорциум по геномным вариациям, 2005, 2008). Этим популяциям присваивается код на основании групп языковой принадлежности (индоевропейская, IE; дравидийская,DR; тибето-бирманская, ТВ; южно-азиатская, АА), затем на основании географической зоны (северная,N; южная, S; восточная, Е; западная, W; центральная, С; северо-восточная NE) и этнической принадлежности (каста LP; род IP; религиозная группа, SP). Описание каждой популяции можно найти в проведенном ранее исследовании. Популяция OG-W-IP1 известного африканского происхождения была включена в качестве внешней группы. 3) 96 не состоящих в кровном родстве мужчин, пациентов с НАРЕ, индоевропейского происхождения были отобраны в SNM (Sonam Norbu Memorial) госпитале (г. Лех (высота 3500 м), штат Джамму и Кашмир, Индия). НАРЕ диагностировали на основании стандартных критериев, что включало оценку начала типичных симптомов вследствие пребывания на большой высоте, в том числе кашля и одышки в состоянии покоя, отсутствия инфекции, наличия легочных хрипов и цианоза, исчезновения симптомов и признаков в течение 3 суток с начала лечения путем предоставления дополнительного кислорода и соблюдения постельного режима (Ahsan и др., 2004). Рентгенографическое обследование грудной клетки показало присутствие инфильтрата, указывающего на отек легких, что подтвердило наличие данного заболевания. После выздоровления пациентов с НАРЕ обследовали, чтобы исключить наличие любых предшествующих заболеваний сердца и легких. Помимо этого при проведении исследований авторы изобретения использовали 96 образцов ДНК жителей Леха, не состоящих в кровном родстве. Отбор генов и SNP Была отобрана подгруппа из 30 генов, которые по данным более раннего исследования демонстрировали различия в экспрессии. В среднем из dbSNP (базы данных по SNP) были отобраны от 6 до 8 целевых SNP на один ген на основании расстояния между маркерами, гетерозиготности, частоты встречаемости и их валидационного статуса в различных популяциях. Целевые SNP из СЕРН-популяции идентифицировали, используя алгоритм селекции Tagger (www.broad.mit.edu/mpg/tagger/) с попарным мечением при отсечении для r2 (коэффициента корреляции), составляющем 0,8. Они в основном представляли собой подтвержденные SNP с описанной гетерозиготностью. Генотипирование SNP и качественный контроль Для генотипирования использовали по 250 нг геномной ДНК на один образец (5 мкл раствора образца ДНК в концентрации 50 нг/мкл), применяя платформу BeadArray и анализ GoldenGate от Illumina в соответствии с протоколом производителя. Обработку начальных данных по интенсивности гибридизации, кластеризацию и определение генотипа (genotype calling) осуществляли, используя относящуюся к генотипированию независимую часть программ (genotyping module) в пакете программ BeadStudio (вер-6 023630 сия 3). Определение генотипа выполняли, используя независимую часть GenCall пакета программ BeadStudio. Кластеры генотипов, сформированные с использованием GenCall для каждого SNP-содержащего локуса, редактировали вручную после визуального просмотра кластеризации на двумерном графике.SNP, которые не соответствовали HWE (равновесию Харди-Вайнберга) на уровне значимости 1% (Р 0,01) более чем в 90% популяций, удаляли из окончательного набора данных. По результатам последующего анализа были исключены 17 SNP, поскольку 5 из них не соответствовали HWE, a 12 не были полиморфными в генотипированных образах. С использованием платформы BeadArray от lllumina (http://iqvbrowser.igib.res.in) в образцах VPK, а также в IGVC-панели были генотипированы в общей сложности 158 SNP из 30 генов, которые по данным предыдущего исследования авторов изобретения демонстрировали различия в экспрессии, и 2060SNP, которые были использованы для популяционной стратификации. Генотипирование rs480902 и rs479200 в образцах пациентов с НАРЕ и уроженцев гор проводили,используя анализ удлинения праймера на одно основание (набор ddNTP (дидезоксинуклеозидтрифосфатов) для удлинения праймеров SNaPshot, Applied Biosystems) на генетическом анализатореABI Prism 3100 после ПЦР-амплификации. Данные о генотипах SNP в гене EGLN1 из 52 популяций брали из HGDP-данных по SNPгенотипированию (Стэнфорд), полученных на чипах 650Y от Illumina. Данные о генотипах, а также круговую диаграмму, отображающую частоту встречаемости rs480902 по всем различным популяциям,(фиг. 5b) брали из браузера для просмотра данных HGDP-отбора (http://hqdp.uchicaqo.edu/cqibin/obrowse/HGDP/). Высоту места проживания каждой из популяций из HGDP-панели брали из проекта Google Планета Земля (Google Earth), используя координаты широты и долготы из HGDP-панели. Количественный ПЦР-анализ Для количественного определения различия в экспрессии EGLN1 среди разных типов пракрити проводили TaqMan-ПЦР, в которой используется специально разработанный анализ с применениемTLDA (чипа "низкой плотности" TaqMan; англ. TaqMan Low Density Array) (Applied Biosystems), на приборе ABI 7900. Статистический анализ Популяционная стратификация и установление генетической гомогенности исследуемой популяции Для анализа популяционной стратификации использовали данные изменчивости для 2060 SNP, не связанных с локусом EGLN1, которые были генотипированы в 24-х популяциях в Индии как часть проекта Индийского консорциума по геномным вариациям, а также в 96 аюрведических образцах. Для детекции популяционной стратификации среди IGVC- и исследуемой популяции использовалиEIGENSTRAT (Price и др., 2006). Данные о генотипах для 2060 SNP из 24-х популяций в Индии брали из данных Индийского консорциума по геномным вариациям (Фаза II). Частоты встречаемости аллелей и генотипов рассчитывали методом подсчета генов (gene-counting method). Отклонения от HWE тестировали, используя точный критерий Фишера. Распределения частот встречаемости аллелей среди групп с разными конституциями сравнивали, используя точный критерий Фишера, выполненный в R. Точный тест Фишера также выполняли для оценки взаимосвязи генотипа и аллелей с НАРЕ. Распределение аллеля сравнивали у пораженных болезнью и здоровых индивидов, используя точный критерий Фишера (FET) и считая значение р 0,05 значимым. Коррекцию для множественного тестирования проводили, используя метод FDR (контроля ложных эффектов; англ. false discovery rate). Для изучения корреляции 'высоты' с различными SNP в EGLN1, генотипированными в IGVC- иHGDP-популяциях, использовали корреляцию рангов Кендалла. Диагностические наборы Согласно данному изобретению также предложен диагностический набор, содержащий последовательности, имеющие ID NO:5, 6, 7, 8, 9, 10. Наборы по настоящему изобретению могут содержать одну или более пар аллель-специфичных олигонуклеотидов, гибридизующихся с различными полиморфными формами. В некоторых наборах предложены аллель-специфичные олигонуклеотиды, иммобилизованные на подложке. Например, одна и та же подложка может содержать аллель-специфичные олигонуклеотидные зонды для детекции, по меньшей мере, выявленного полиморфизма в гене EGLN1. Возможные дополнительные компоненты набора включают, например, рестрикционные ферменты, обратную транскриптазу или полимеразу, нуклеозидтрифосфаты в качестве субстратов, средства, используемые для введения метки (например, конъюгат авидин-фермент, и субстрат для фермента, и хромоген, если метка представляет собой биотин), и соответствующие буферы для обратной транскрипции, ПЦР или реакций гибридизации. Набор также может содержать инструкцию для осуществления способов по настоящему изобретению. В инструкции просто описываются воплощения изобретения, изложенные в данном описании. Нуклеиновокислотные векторы Вариантные гены можно экспрессировать с использованием экспрессирующего вектора, в котором вариантный ген функционально связан со своим природным или другим промотором. Обычно промотор представляет собой эукариотический промотор для экспрессии в клетке млекопитающего. Регулирующие транскрипцию последовательности обычно включают в себя гетерологичный промотор и возможно энхансер, которые узнаются хозяином. Выбор соответствующего промотора, например промоторов trp(триптофанового оперона), lac (лактозного оперона), фаговых промоторов, промоторов гликолитических ферментов и промоторов тРНК, зависит от выбранного хозяина. Кроме того, можно использовать имеющиеся в продаже экспрессирующие векторы. Подходящие клетки хозяина включают клетки бактерий,таких как Е. coli, дрожжей, мицелиальных грибов, клетки насекомых, клетки млекопитающих, обычно иммортализованные, например, мышиные клеточные линии, клеточные линии СНО (яичника китайского хомячка), человека и обезьяны и их производные. Предпочтительные клетки хозяина способны осуществлять процессинг продукта вариантного гена с получением соответствующего зрелого полипептида. Следующие далее примеры приведены для иллюстрации настоящего изобретения и их не следует рассматривать как ограничивающие объем настоящего изобретения. Примеры Пример 1. Эндофенотипирование Опросник (рег.авторского права SW-2284/2005, дата регистрации 13 мая 2005 г.) для проведения клинического фенотипирования был разработан на основе аюрведической литературы по фенотипам и методам оценки пракрити (анализ основных конституций), подробное описание которых приведено в опубликованной ранее статье. Фенотипическая классификация, в целом, учитывает параметры, связанные с анатомическими особенностями, подобные телосложению, скелету, размеру и симметрии частей тела, физиологии, физической выносливости и способностям. Проводили скрининг 850 индивидов на предмет анализа конституции их тела (пракрити). Среди них были выявлены 96 индивидов с преобладающей в пракрити дошей, в том числе ватой (39), питтой(29) и капхой (28), и они были отобраны для сбора образцов. Эти индивиды из возрастной группы 18-40 лет (средний возраст 234 года) имели индоевропейское происхождение и включали приблизительно одинаковое число представителей обоих полов (1). Пример 2. Отбор субъектов и сбор образцов Идентификацию индивидов с преобладающими типами пракрити проводили два врача Аюрведы. Чтобы избежать какого-либо искажения результатов наблюдений вследствие популяционной стратификации, исследование проводили на больших популяциях людей, говорящих на индоевропейских языках,преимущественно из Северной Индии. Предварительная оценка пракрити была проведена в общей сложности на 850 добровольцах, примерно поровну двумя практикующими врачами, с использованием субъективной оценки и скринингового опросника. Кроме того, независимо составляли укороченный список индивидов, отобранных для подробного фенотипирования. Эти два врача-клинициста производили обмен индивидами, внесенными в укороченный список, и подробно оценивали их в отношении пракрити,используя опросник. Сюда входили примерно 120 индивидов с преобладающим типом пракрити и 200 индивидов со смешанным типом пракрити. Примерно 80% составило соответствие в оценке пракрити двумя врачами-клиницистами. После этого были выявлены и включены в исследование 96 здоровых, не состоящих в кровном родстве этнически равноценных индивидов с преобладанием или ваты (39 индивидов), или питты (29), или капхи (28), с равным представительством обоих полов (n = 48 в каждом случае) и принадлежностью к возрастной группе 18-40 лет (средний возраст 234 года). Образцы периферической крови отобранных индивидов собирали с использованием стандартных методик, следуя этическим нормам Индийского совета по медицинским исследованиям (Индия), и с осознанного согласия добровольцев. Отбор образцов проводили с разрешения ведомственного комитета по биоэтике (IBC). За три часа до отбора образцов всем добровольцам предоставляли одинаковое питание без какого-либо промежуточного приема пищи, напитков или курения. Следили за тем, чтобы субъект не оказался больным или не находился под воздействием какого-либо лекарственного средства. Кровяное давление, пульс и менструальный цикл, если он имелся, также регистрировали. До проведения исследования с использованием этих отобранных образцов образцы кодировали для сохранения их анонимности. Пример 3. Выделение геномной ДНК Геномную ДНК выделяли из лейкоцитов периферической крови отобранных индивидов чистых типов пракрити, используя модифицированную методику высаливания (Miller и др., 1988). 1) Кислый цитратный буфер на основе декстрозы (ACD) 0,48 г лимонной кислоты,1,32 г цитрата натрия,1,47 г глюкозы. Растворить в воде в конечном объеме 100 мл. Стерилизовать в автоклаве и хранить при 4 С. 2) Буфер для лизиса RBC (эритроцитов) (10) Растворить в 100 мл дистиллированной воды, стерилизовать в автоклаве и хранить при 4 С. Рабочее разведение (1). На 500 мл буфера для лизиса RBC (1 х): 50 мл буфера для лизиса RBC (10) + 450 мл стерилизованной в автоклаве воды. 3) Буфер для лизиса ядер (NLB) 10 мМтрис-HCl, 400 мМ NaCl,2 мМ Na2-EDTA (pH 8,0) (стерилизовали в автоклаве и хранили при комнатной температуре). Для 400 мл: Конечный объем подвести до 400 мл (стерилизовали в автоклаве и хранили при комнатной температуре). 4) Раствор протеиназы К (20 мг/мл) 20 мг протеиназы K растворяли в 1 мл стерилизованной в автоклаве дистиллированной воды. Хранили при 4 С. 5) 10%-ный SDS (додецилсульфат натрия) Для 100 мл: 10 г SDS растворяли в стерилизованной в автоклаве дистиллированной воде. Конечный объем подводили до 100 мл (хранили при комнатной температуре). 6) 6 M насыщенный раствор NaCl NaCl - 35,064 г. Растворяли в дистиллированной воде и конечный объем подводили до 100 мл. 7) ТЕ-буфер (200 мл) 10 мМ трис (рН 8,0),1 мМ EDTA (рН 8,0). Стерилизовали в автоклаве и хранили при 4 С. Выделение ДНК из кровиОтобрать по 2-10 мл крови и перенести в соответственно помеченные пробирки.Удалить из крови плазму центрифугированием при 2000 об./мин в течение 10 мин.К оставшемуся объему крови добавить буфер для лизиса RBC (1), подводя объем до 50 мл.Перемешать суспензию, переворачивая пробирки несколько раз, пока она не станет полупрозрачной.Выдержать ее при комнатной температуре в течение приблизительно 20 мин со слабым встряхиванием, пока полностью не завершится действие буфера для лизиса. После этого отцентрифугировать лизированную кровь при 2500 об./мин в течение 10 мин при комнатной температуре. Слить супернатант в гипохлорит. К осадку центрифугирования добавить 15 мл буфера для лизиса RBC и смешать путем недолгого перемешивания на вортексе. Еще раз отцентрифугировать при 1000 об./мин в течение 10 мин при комнатной температуре. Слить супернатант в гипохлорит. К осадку центрифугирования добавить 12 мл буфера для лизиса ядер и перемешать на вортексе. Добавить 0,8 мл 10%-ного SDS и 50 мкл раствора протеиназы К (20 мг/мл) и хорошо перемешать. Инкубировать пробирки при температуре 65 С в течение 2 ч в водяной бане. Добавить 4 мл насыщенного раствора NaCl (6 M) и энергично встряхивать в течение 15 с. Незамедлительно отцентрифугировать пробирку при 3500 об./мин в течение 30 мин при комнатной температуре. Аккуратно отобрать супернатант, не затрагивая осадка центрифугирования, добавить 2 объема (то есть довести объем до 50 мл) абсолютного этанола (то есть 100%-ного) и оставить при комнатной температуре. Осадить (ppt) ДНК, очень медленно переворачивая 10-20 раз (или оставить на ночь при -20 С). Перенести осажденную ДНК, используя наконечник пипетки, в микроцентрифужную пробирку, содержащую 1 мл 70%-ного этанола. Пробирки недолго перемешать на вортексе и отцентрифугировать при 13000 об./мин в течение 10 мин. Аккуратно извлечь супернатант, не затрагивая осадка центрифугирования. Осадок центрифугирования высушить на воздухе в течение 1-2 ч и ресуспендировать в 0,5-1 мл ТЕбуфера (трис+EDTA), рН = 8,0. Растворить ДНК, оставив ее при температуре 65 С на 2 ч, и хранить при температуре -20 С. Пример 4. Конструирование и синтез праймеров (ПЦР- и SNaPshot-праймеров) Пары ПЦР-праймеров, а также SNaPshot-праймеры конструировали для выбранных SNP, используя праймер 3 и программное обеспечение Assay Designer от Sequenom. Запланированные праймеры были синтезированы в TCGA (Центре прикладной геномики; англ. The Centre for Genomic Applications). Пример 5. Полимеразная цепная реакция (ПЦР) Реагенты: 1. 25 мМ MgCl2 (100 мл) Сначала приготовить 1 M MgCl2 (50 мл).W = (50203,311)/1000 = 10,1655 г. Растворить 10,1655 г MgCl2 в 50 мл Milli Q, затем стерилизовать автоклавированием. Для 25 мМ MgCl2 (100 мл): 25 мМ 100 мл = 11000 мМX мл,X мл = 2500/1000 = 2,5 мл. Затем смешать 2,5 мл 1 M MgCl2 и 97,5 мл стерилизованной автоклавированием Milli Q. 2. 10 буфер для ПЦР (100 мл) 100 мМ трис рН 9,0,500 мМ KCl, 0,1% желатина.W = (1121,4450)/1000 = 6,07 г. Сначала 6,07 г триса растворяют в 40 мл стерилизованной автоклавированием воды, очищенной с использованием системы Elix. Подвести рН до 9,0 и довести объем до 50 мл.W = (15074,55)/1000 = 3,72 г. Растворить 3,72 г KCl в 50 мл стерилизованной автоклавированием воды, очищенной с использованием системы Elix.(c) 0,1% желатина Для 100 мл 10 буфера для ПЦР: 1 М трис= 10 мл,1 M KCl = 50 мл,желатин = 100 мг (первоначально растворенный в воде). Затем довести объем до 100 мл и стерилизовать автоклавированием. 3. 25 мМ каждого из dNTP (дезоксинуклеозид-трифосфатов) (50 мл) Концентрированный раствор каждого из dNTP = 100 мМ. Рабочая концентрация = 25 мМ. Для 50 мл 25 мМ раствора каждого из dNTP: N1V1 =N2V2 100 мМV1(мкл) = 2 мМ 50000 мкл V1 =(250000)/100 V1 = 1000 мкл или 1 мл. Так добавить 1 мл dATP, 1 мл dTTP, 1 мл dGTP, 1 мл dCTP и 46 мл воды Milli Q. 4. Taq ДНК-полимераза (Genei, Bangalore; 3 ед/мкл) 5. Локус-специфные праймеры Для стандартной ПЦР-реакции в объеме 10 мкл. ПЦР-реакции проводили с начальной денатурацией при 94 С в течение 5 мин, затем следовали 30 циклов денатурации при 94 С по 30 с и стадия удлинения при 72 Св течение 45 с. Стадию отжига проводили в течение 30 с, а температура отжига составляла 56 С. Реакцию завершали, выполняя конечную стадию удлинения при 72 С в течение 10 мин. Контроль в геле 1. 50 ТАЕ (рН 8.5) Для 1000 мл: трис: 242 г, EDTA: 37,2 г,ледяная уксусная кислота: 56 мл. Довести объем до 1000 мл. 2. Раствор бромистого этидия 1000 концентрированный раствор (0,5 мг/мл): 50 мг EtBr, 100 мл H2O. Защищать от света. 3. 10 Краситель для нанесения образцов 50% глицерина,0,25% бромфенолового синего,0,25% ксиленцианола. Проверку ПЦР-продукта выполняли на 2%-ном агарозном геле, используя лэддер длиной 100 п.о. Если происходила амплификация конкретной зоны, то процесс продолжали. Пример 6. Протокол очистки ПЦР-продукта с использованием ПЭГ (полиэтиленгликоля) ПЭГ/раствор ацетата натрия: Довести до конечного объема 50 мл, используя H2O MilliQ. Два объема раствора ПЭГ/NaOAc добавляли к ПЦР-продукту, тщательно перемешивали на вортексе и инкубировали в течение 10 мин при комнатной температуре. ДНК осаждали центрифугированием при 3200 об./мин в течение 30-60 мин в зависимости от размера продукта амплификации. Супернатант удаляли, переворачивая ПЦР-планшет на бумажную салфетку и центрифугируя при 500 об/мин. Осадок центрифугирования дважды промывали двумя объемами 70%-ного этанола, используя центрифугирование при 3200 об./мин в течение 10 мин. Этанол полностью удаляли и осадок центрифугирования сушили на воздухе. Пример 7. Генотипирование SNP с использованием SNaPshot-метода Авторы изобретения проводили реакции удлинения праймера на одно основание (набор ddNTP для удлинения праймеров SNaPshot, Applied Biosystems). Кратко, SNaPshot-реакцию проводили с использованием очищенного с помощью ПЭГ ПЦР-продукта, 1 мкл генотипирующего праймера (2 пг/мкл), 0,5 мкл готовой реакционной смеси SNaPshot, 0,8 мкл 5 буфера для разведения (200 мМ трис, рН 9,0; 5 мМMgCl2) и воды MilliQ, для доведения объема до 5 мкл. ПЦР проводили в следующих условиях: 94 С в течение 3 мин, затем 40 циклов при 96 С по 10 с, при 50 С по 5 с и при 60 С по 30 с. Неинкорпорированные ddNTP среди продуктов SNaPshot-реакции переваривали посредством инкубации образцов с 0,25 единицами щелочной фосфатазы кишечника теленка (CIP) при 37 С в течение 1 ч, затем посредством инкубации с CIP при 72 С в течение 15 мин. По 2 мкл продуктов SNaPshot-реакции смешивали с 8 мкл Hi-Di (высокоочищенного деионизованного) формамида и вносили в генетический анализатор 3100. Пример 8. Количественный ПЦР-анализTAQMAN-ПЦР, в которой используется специально разработанный анализ с применением TLDA(Applied Biosystems), проводили на приборе ABI 7900. Каждый эксперимент проводили в трех повторах. РНК из всех образцов подвергали обратной транскрипции с получением кДНК, используя набор архивов кДНК высокой емкости (High Capacity cDNA Archive kit; Applied Biosystems, Foster City, CA), следуя рекомендованным производителем протоколам. кДНК амплифицировали, используя универсальную мастер-смесь TaqMan для ПЦР (Applied Biosystems, Foster City, CA). Следили за тем, чтобы количество матрицы кДНК, добавленное в каждую реакционную смесь, находилось в определенных пределах с це- 11023630 лью получения относительно узкого диапазона Ct (порогового цикла; англ. cycle threshold), определяемого в качественном контрольном измерении кДНК 18S pPHK. Пример 9. Анализ GoldenGate от Illumina Платформа от Illumina подходит для генотипирования сотен SNP в одной многоканальной системе с тысячами образцов (Fan и др., 2006). Этот анализ позволяет достигать высокой степени мультиплексирования локусов (loci multiplexing), при этом 1536 полиморфизмов можно изучать одновременно в 96 образцах или кратном 96 количестве образцов. Концентрацию ДНК в образцах оценивали, используя метод PicoGreen, и образцы разбавляли до 50 нг/мкл. На один образец использовали аликвоту 5 мкл (250 нг) разбавленной ДНК. После отбора SNP все образцы SNP направляли в Assay Design Tool (устройство для разработки анализа) от Illumina для выставления баллов в отношении разработки ОРА (весь пул олигонуклеотидов).SNP-баллы были выставлены с помощью Illumina, и значения SNP-баллов лежали в диапазоне от 0 до 1,1. SNP-балл отражает возможность разработки успешного анализа. В настоящем исследовании SNP-балл 0,4 был выбран в качестве нижнего предела. SNP, не достигшие этого балла, заменяли на близлежащие SNP и еще раз направляли в Illumina для выставления баллов. Окончательный список SNP с баллом 0,4 затем был направлен в Illumina для разработки ОРА и синтеза. Краткое введение в данный метод представлено ниже. Для каждого SNP синтезируют три олигонуклеотида (олига): два аллель-специфичных олига (ASO), которые узнают аллели SNP, и один локусспецифичный олиг (LSO) сразу за этим SNP. Кроме того, последовательности ASO и LSO содержат целевые последовательности для набора универсальных праймеров (Р 1 и Р 2 для двух ASO и РЗ для LSO),при этом каждый LSO также содержит конкретные адресные последовательности (illumicode), комплементарные последовательностям, присоединенным к гранулам. Все олиги, с помощью которых уточняют набор SNP, синтезируются и объединяются в общий пул в Illumina. Для проведения анализа этот объединенный набор олигов (ОРА) гибридизуют одновременно с геномной ДНК, представленой в одном(й) образце/реакционной лунке. После осуществления реакций удлинения аллель-специфичного праймера и лигирования добавляют набор флуоресцентно меченных универсальных праймеров (Р 1 и Р 2, меченных Су 3 и Су 5, соответственно) и проводят ПЦР, генерируя множественные меченые продукты амплификации, представляющие сотни различных SNP. Затем эти флуоресцентные продукты объединяют с гранулами на Sentrix Array Matrix (матрице чипа Sentrix) (SAM). Адресные последовательности в продуктах ПЦР-амплификации гибридизуются с соответствующими им последовательностями на гранулах, и флуоресценцию от каждой гранулы измеряют количественно, получая сигнал, ассоциированный с конкретной адресной последовательностью. Каждая адресная последовательность указывает на конкретный локус, и присутствие Су 3, Су 5 или обоих сигналов на данном типе гранул указывает на генотипы АА, ВВ или АВ. Перед проведением эксперимента в 96-луночный ПЦР-планшет вносили по 250 нг ДНК на один образец (5 мкл раствора в концентрации 50 нг/мкл). Чтобы убедиться в воспроизводимости анализа, 10% образцов готовили в двух повторах. Эти 96-луночные ПЦР-планшеты обрабатывали согласно рекомендациям. После гибридизации проводили сканирование SAM, используя ридер для чипов с гранулами(beadarray reader) Beadstation 500. Интенсивности сигналов гибридизации с ридера для чипов с гранулами применяли для обработки данных, кластеризации и определения генотипа, используя относящуюся к генотипированию независимую часть программ в пакете программ BeadStudio (версия 3). Определение генотипа выполняли, используя независимую часть GenCall пакета программ BeadStudio. Кластеры генотипов, сформированные для каждого локуса SNP с использованием GenCall, редактировали вручную после визуального просмотра кластеров на двумерном графике. Все кластеры генотипов проверяли и корректировали вручную; был установлен порог для GenTrain-балла 0,25, чтобы считать SNP успешно генотипированным (Fan и др., 2006). Все повторы, использованные для проверки точности установления генотипа, показали уровень совпадения 99%. Большинство образцов хорошо зарекомендовали себя и показали высокий уровень идентификации (call rate) среди всех SNP, генотипированных в диапазоне 85100% (средний уровень идентификации 0,990,02). Пример 10. Статистический анализ Частоты встречаемости аллелей и генотипов рассчитывали методом подсчета генов. HWE рассчитывали, используя точный критерий Фишера. Распределения частот встречаемости аллелей среди групп с разными пракрити сравнивали, используя программу Plink. Коррекцию для множественного тестирования проводили, используя метод FDR. Точный тест Фишера также выполняли для оценки взаимосвязи генотипа и аллелей с НАРЕ. Распределение аллеля сравнивали у пораженных болезнью и здоровых индивидов, используя точный критерий Фишера (FET) и считая значение р 0,05 значимым. Из панели образцов из 64 генов авторы изобретения идентифицировали два контрольных генаVBA для Microsoft Excel), в качестве внутреннего контроля для измерения относительной экспрессииEGLN1 среди разных типов конституции. Кратко, в этом методе идентифицируют наиболее стабильные контрольные гены из панели генов-кандидатов. С использованием geNORM рассчитывают меру стабильности генной экспрессии M для контрольного гена как среднюю попарную вариацию V для этого гена со всеми другими тестируемыми контрольными генами. Поэтапное исключение генов с наиболее высокими величинами M позволяет ранжировать контрольные гены-кандидаты в соответствии со стабильностью их экспрессии. Затем определяют нормирующий множитель генной экспрессии для каждого образца на основе среднего геометрического определенного пользователем числа генов домашнего хозяйства. На основании низких или высоких средних величин Ct делали заключение о стимулирующей регуляции или об угнетающей регуляции, соответственно. Сравнение различий в экспрессии EGLN1 относительно генотипов rs479200 проводили, используя односторонний критерий Стьюдента. Для изучения корреляции "высоты" с "частотой встречаемости Т" rs480902, a также с SNP в EGLN1, генотипированными в обеих IGVC- и HGDP-популяциях, авторы изобретения использовали корреляцию рангов Кендалла. Преимущества Настоящее изобретение полезно для выявления индивидов, которые могут быть подвержены риску развития НАРЕ, когда они путешествуют или направляются в высокогорные места. Данная информация может быть полезна для молекулярной диагностики как на генетическом уровне, так и на уровне экспрессии, для предсказания адаптируемости индивида к большой высоте, сопротивляемости, восприимчивости к НАРЕ и другим заболеваниям, в которые вовлечена гипоксия, например к сердечнососудистым заболеваниям, прогрессированию рака, астме, нейродегенерации, ишемии, инсульту, болезням печени, заболеваниям кожи, артриту и т.д., а также восприимчивости к ингибиторам PHD2. ФОРМУЛА ИЗОБРЕТЕНИЯ 1. Биомаркер, пригодный для прогнозирования предрасположенности индивидуума к высокогорной адаптации и высокогорному отеку легких (НАРЕ), имеющий однонуклеотидный полиморфизм С/Т в положении 27 в SEQ ID NO: 1 и Т/С в положении 27 в SEQ ID NO: 2 гена EGLN1. 2. Биомаркер по п.1, имеющий SEQ ID NO: 2, где частота встречаемости "С"-аллеля rs480902 y людей, у которых развивается высокогорный отек легких, существенно (значение р 0,05) выше таковой у людей, проживающих на большой высоте. 3. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "С"-аллеля rs479200 y людей, проживающих на большой высоте, существенно (значение р 0,05) выше таковой у людей, проживающих на незначительной высоте. 4. Биомаркер по п.1, имеющий SEQ ID NO: 2, где частота встречаемости "СС"-генотипа rs480902 y людей, у которых развивается высокогорный отек легких, существенно (значение р 0,05) выше таковой у людей, проживающих на большой высоте. 5. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "Т"-аллеля rs479200 y людей, у которых развивается высокогорный отек легких, существенно (значение р 0,05) выше таковой у людей, проживающих на большой высоте. 6. Биомаркер по п.1, имеющий SEQ ID NO: 1, где частота встречаемости "ТТ"-генотипа rs479200 y людей, у которых развивается высокогорный отек легких, существенно (значение р 0,05) выше таковой у людей, проживающих на большой высоте. 7. Биомаркер по п.1, имеющий SEQ ID NO:2, где частота встречаемости "Т"-аллеля rs480902 y людей, проживающих на большой высоте, существенно (значение р 0,05) выше таковой у людей, проживающих на незначительной высоте. 8. Праймер, пригодный для амплификации биомаркера по пп.1-7, имеющий последовательность,выбранную из группы, состоящей из: которая является последовательностью SNaPshot-праймера. 9. Способ получения биомаркера по п.1, включающий:a) выделение геномной ДНК из субъекта, являющегося человеком;c) амплификацию плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью SEQ IDe) амплификацию минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью SEQ IDf) определение SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO: 7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно. 10. Способ детекции высокогорной адаптации и предрасположенности индивидуума к высокогорному отеку легких, включающий стадии:a) выделения геномной ДНК из субъекта, являющегося человеком;c) амплификации плюс-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 3, с использованием праймеров, синтезированных на стадии (b), для получения биомаркера с последовательностью SEQ IDe) амплификации минус-цепи интрона 1 гена EGLN1, имеющей SEQ ID NO: 4, с использованием праймеров, синтезированных на стадии (d), для получения биомаркера с последовательностью SEQ IDf) определения SNP у пациентов с НАРЕ и у уроженцев горной местности с использованием SNaPshot-праймеров с SEQ ID NO: 7 и 10 для SNP ID rs479200 и SNP ID rs480902 соответственно;g) подсчета частот встречаемости ТТ-, ТС- и СС-генотипов в популяциях со стадии (f) для установления взаимосвязи данных генотипов с высокогорной адаптацией и высокогорным отеком легких;h) прогнозирования и статистического анализа различий в распределении аллельных вариантов (Т,С) в популяции, где ТТ-генотип rs479200 и СС-генотип rs480902 в гене EGLN1 связаны с высоким риском высокогорного отека легких, а СС-генотип rs479200 и ТТ-генотип rs480902 в гене EGLN1 связаны с низким риском высокогорного отека легких. 11. Набор для детекции высокогорной адаптации и предрасположенности к высокогорному отеку легких у человека, содержащий: 1) праймеры, имеющие SEQ ID NO: 5, 6, 7, 8, 9, 10; 2) подходящие реагенты; 3) техническую документацию. 12. Применение биомаркеров для прогнозирования предрасположенности индивидуума к высокогорной адаптации и высокогорному отеку легких (НАРЕ), имеющих однонуклеотидный полиморфизм С/Т в положении 27 в SEQ ID NO: 1 и Т/С в положении 27 в SEQ ID NO: 2 гена EGLN1.
МПК / Метки
МПК: C12Q 1/68
Метки: легких, биомаркер, адаптации, отека, высокогорной, детекции, высокогорного
Код ссылки
<a href="https://eas.patents.su/30-23630-biomarker-dlya-detekcii-vysokogornojj-adaptacii-i-vysokogornogo-oteka-legkih.html" rel="bookmark" title="База патентов Евразийского Союза">Биомаркер для детекции высокогорной адаптации и высокогорного отека легких</a>
Предыдущий патент: Огнестойкий строительный раствор
Следующий патент: Замещенные 5-фтор-1н-пиразолопиридины и их применение
Случайный патент: Элемент из шлаковой ваты, обладающий повышенной стойкостью к действию огня, и воздуховод, включающий указанный элемент