Способ прогнозирования белок-белковых взаимодействий
Формула / Реферат
1. Способ прогнозирования белка или полипептида, взаимодействующих с конкретным белком или полипептидом (A), который включает следующие стадии:
1) декомпозиция-перегруппировка последовательности аминокислот белка или полипептида (A) в серию олигопептидов, обладающих заданной длиной в качестве информации о последовательности;
2) поиск в базе данных по последовательностям аминокислот такого белка или полипептида (C), который включал бы в себя последовательность аминокислот для каждого члена данной серии или для белка или полипептида (D), содержащего последовательность аминокислот, гомологную последовательности аминокислот каждого члена данной серии;
3) выполнение локальной линейной сверки сходства указанного белка или полипептида (A) с отобранным белком или полипептидом (C) или с отобранным белком или полипептидом (D) и
4) прогнозирование того, является ли установленный белок или полипептид (C) и/или белок или полипептид (D) белком или полипептидом (B), который взаимодействует с белком или полипептидом (A), на основе результатов локальной линейной сверки последовательностей аминокислот и на основе значения, подсчитанного, исходя из частоты встречаемости аминокислот и /или частоты встречаемости вышеуказанных олигопептидов в вышеупомянутой базе данных по последовательностям аминокислот.
2. Способ по п.1, где олигопептид состоит из 4-14 аминокислот.
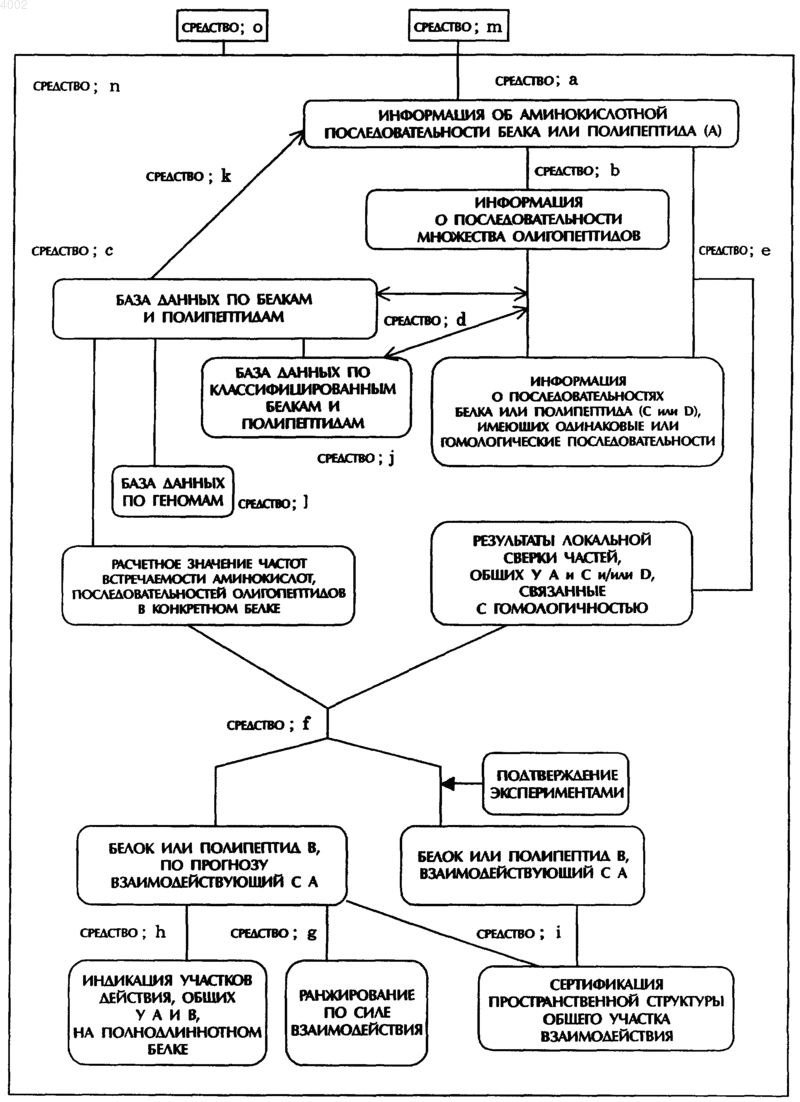
3. Среда для записи, несущая программу для прогнозирования белка или полипептида (B), который взаимодействует с конкретным белком или полипептидом (A), при этом данная среда для записи характеризуется включением в свой состав, по крайней мере, следующих средств a) - f):
a) средство для ввода информации о последовательности аминокислот в белке или полипептиде (C) и запоминания этой информации;
b) средство перегруппировки-декомпозиции вышеуказанной информации в серию олигопептидов, имеющих заданную длину в качестве информации о последовательности, и соответственно средство для запоминания полученной информации о последовательности;
c) средство хранения введенной базы данных по белкам;
d) средство для доступа к хранимой базе данных по белкам и для установления белка или полипептида (C), имеющего аминокислотную последовательность вышеуказанного олигопептида, или белка или полипептида (D), имеющего аминокислотную последовательность, гомологическую аминокислотной последовательности вышеуказанного олигопептида, и средство хранения и подсчета установленного результата;
e) средство для выполнения локальной линейной сверки вышеуказанного белка или полипептида (A) с установленным белком или полипептидом (C) или с белком или полипептидом (D) и средство для хранения и подсчета результата и
f) средство для получения результирующего значения частоты встречаемости аминокислоты и/или частоты встречаемости вышеназванного олигопептида по базе данных по белкам с последующим указанием индекса для прогноза белок-белковых взаимодействий на основе этого результирующего значения и результирующего значения вышеупомянутой локальной линейной сверки и средство для запоминания и вывода на дисплей результата и соответственно установленного белка или полипептида (B), который взаимодействует с белком или полипептидом (A).
4. Среда по п.3 дополнительно содержащая, по крайней мере, следующие средства:
g) средство для ранжирования силы белок-белковых взаимодействий у установленных белков или полипептидов (B), основывающегося на индексах, подсчитанных на основе результирующего значения локальной линейной сверки и результирующего значения частоты встречаемости аминокислоты и/или частоты встречаемости олигопептида в базе данных по белкам для случая, когда существует более чем один белок или полипептид (B) из тех, что были установлены, и средство для запоминания и демонстрации данного результата.
5. Среда по п.3 или 4 дополнительно содержащая, по крайней мере, следующее средство:
h) средство для демонстрации полной длины аминокислотных последовательностей вышеуказанного белка или полипептида (A) и вышеуказанного белка или полипептида (B), который был установлен, с последующим индикаторным обозначением месторасположения частичной последовательности, которая подлежит линейной сверке, в полнодлиннотной последовательности для случая, когда частичные последовательности аминокислот сверяются посредством локальной линейной сверки между белком или полипептидом (A) и белком или полипептидом (B).
6. Среда по любому из пп.3, 4 и 5 дополнительно включающая в свой состав, по крайней мере, следующее средство:
i) средство для расчета модели пространственной структуры для случая, когда пространственная структура вышеуказанного белка или полипептида (A) или вышеуказанного белка или полипептида (B), который был установлен, является известной, или для случая, когда моделирование по гомологии дает возможность построить модель пространственной структуры, с последующим выводом на дисплей этой структуры частичных аминокислотных последовательностей, которые выверяются по пространственной структуре путем локальной линейной сверки белка или полипептида (A) и у белка или полипептида (B).
7. Среда по любому из пп.3-5 или 6, дополнительно включающая в свой состав, по крайней мере, следующее средство:
j) средство классификации и запоминания белков в базе данных по белкам для того, чтобы сузить зону поиска.
8. Среда по любому из предшествующих пунктов, дополнительно включающая в свой состав, по крайней мере, следующее средство:
k) средство для посерийного ввода каждого белка в базу данных по белкам, такого как вышеуказанный белок или полипептид (A).
9. Среда по любому из предшествующих пунктов, дополнительно включающая в свой состав, по крайней мере, следующее средство:
l) средство для хранения базы данных по геномам.
10. Устройство для прогнозирования межбелковых взаимодействий, включающее в свой состав те средства, носителем которых является среда по пп.3-8 или 9.
11. Способ определения белков или полипептидов, способных взаимодействовать друг с другом, который включает идентификацию белка или полипептида (B), взаимодействующего, как это было спрогнозировано на основе применения способа по п.1 или 2, с конкретным белком или полипептидом (A), а затем дальнейшее экспериментальное подтверждение наличия взаимодействия между белком или полипептидом (A) и белком или полипептидом (B).
12. Способ определения белков или полипептидов, взаимодействующих друг с другом, который включает идентификацию белка или полипептида (B), взаимодействующего с конкретным белком или полипептидом (A) согласно прогнозу, сделанному с применением устройства по п.10, а затем экспериментальное подтверждение наличия взаимодействия между белком и полипептидом (A) и белком или полипептидом (B).
13. Белок или полипептид, который установлен способом по п.11 или 12.
14. Способ скрининга соединения, контролирующего взаимодействие конкретного белка или полипептида (A) и белка или полипептида (B), в котором применяется способ по п.1 или 2.
15. Способ скрининга соединения, контролирующего взаимодействие конкретного белка или полипептида (A) и белка или полипептида (B), в котором применяется устройство по п.10.
16. Соединение, полученное способом по п.14 или 15.
17. Соединение, способное контролировать взаимодействие конкретного белка или полипептида (A) и белка или полипептида (B), которое получено путем конструирования фармакосредств (drug design) на основе информации о соединении, полученном способом по п.14 или 15.
18. Олигопептид, содержащий аминокислотную последовательность SEQ ID No:1, который способен контролировать взаимодействие веротоксина 2 (VTII) с Bcl-2.
19. Агент против гибели клеток, содержащий олигопептид, включающий аминокислотную последовательность SEQ ID No:1.
20. Олигопептид, включающий аминокислотную последовательность, гомологную последовательности аминокислот SEQ ID No:1, который способен контролировать взаимодействие VTII и Bcl-2.
21. Полипептид, содержащий аминокислотную последовательность такую же, каъ у олигопептида по п.18 или 20, который способен контролировать взаимодействие VTII и Bcl-2.
22. Способ скрининга, способного контролировать взаимодействие VTII и Bcl-2, в котором используется олигопептид по п.18 или 20 и/или полипептид по п.21.
23. Способ определения нуклеотидной последовательности в олигонуклеотиде, кодирующей олигопептид, который вовлечен во взаимодействие конкретного белка или полипептида (A) и белка или полипептида (B), где используется способ п.1 или 2 или устройство для прогнозирования согласно п.10.
24. Набор серии комбинаций человеческих белков, в отношении которых получен прогноз, что они взаимодействуют друг с другом, с применением способа по п.1 или 2 или с помощью устройства согласно п.10.
25. Способ селекции комбинации белков, взаимодействующих друг с другом, где упомянутое взаимодействие связано с заболеванием; причем указанный способ включает селективный отбор комбинации на основе информации о каком-либо известном белке, который может иметь отношение к этому заболеванию, из набора серии комбинаций по п.24.
26. Набор серии комбинаций белков, которые взаимодействуют друг с другом, где данное взаимодействие относится к заболеванию; при этом каждый член вышеуказанной серии отбирается по способу согласно п.25.
27. Способ скрининга соединения, которое контролирует взаимодействие комбинации белков и/или двух белков, отбираемых из серии комбинаций по п.26.
28. Соединение, полученное способом по п.27.
29. Способ прогнозирования процессируемого участка белка, включающий прогнозирование взаимодействия конкретного белка с ферментом, расщепляющим данный белок, с применением способа по п.1 или 2 или с применением устройства по п.10.
30. Полипептид, содержащий аминокислотную последовательность, которая в соответствии с полученным прогнозом включает участок процессируемого белка, полученный способом п.29, и/или включает частичную последовательность, гомологическую этому процессируемому участку.
Текст
1 Область техники, к которой относится изобретение Настоящее изобретение относится к прогнозу белок-белок взаимодействий, к способу и устройству для этого и к белкам, полученным с применением данного способа и данного устройства. Уровень техники Многие белки выполняют свои функции путем взаимодействия с другими белками или с аналогичными белками. Поэтому объяснение межбелковых взаимодействий представляется важным для развития фармацевтики, бридинга в сельском хозяйстве и т.п. Следует отметить также, что вместе с прогрессом геномного анализа и анализа cDNA различных организмов,включая патогенные микроорганизмы, быстро растет число вновь открываемых генов и кодируемых ими белков, чьи функции неизвестны. Объяснив белок-белковое взаимодействие,можно будет предсказывать функцию белка, чьи функции были неизвестны. К числу стандартных методов, используемых для демонстрации взаимодействия одного белка с другим конкретным белком с целью объяснения их взаимодействия, относится так называемая двухгибридная система (Field, S.Enzymol., 5, 116-124, 1993). Однако эта двухгибридная система представляет собой эксперимент, основанный на скрининге, чьи операции сложны для осуществления и требуют много времени. Кроме того, количество белков, получаемых таким способом, оказывается ниже,чем ожидалось, и дополнительно способ имеет тот недостаток, что результаты его зависят от качества используемой библиотеки cDNA. Другими словами, с этим методом мы рискуем тем,что гена, кодирующего белок, взаимодействующий с данным конкретным белком, может не оказаться в используемой библиотеке cDNA. С другой стороны, белковые базы данных настолько улучшились на основе геномного анализа и cDNA-анализа, что был принят новый метод, при котором белковый комплекс в клетке подвергается непосредственно масс-спектрометрии MALDI-TOF, исходя из чего далее осуществляется поиск по базе данных фрагмента этой аминокислотной последовательностиHuphrey-Smith, I., et al., Electrophoresis, 18, 12171242; Kaufmann, R., 1995, J. Biotechnol., 41, 155176, 1997). Этот метод дает информацию относительно белков, которые образуют комплекс,но не дает никакой информации о межбелковом взаимодействии. И, таким образом, приходится устанавливать экспериментально, какие из белков взаимодействуют друг с другом. Сущность изобретения В одном своем воплощении настоящее изобретение относится к способу прогнозирова 004002 2 ния белка или полипептида (В), взаимодействующего с данным конкретным белком или полипептидом (А), который характеризуется тем,что включает следующие стадии: 1) перегруппировку аминокислотной последовательности белка или полипептида (А) в серию олигопептидов, обладающих заданной длиной в качестве информации о последовательности; 2) поиск в пределах базы данных белков или полипептидов аминокислотных последовательностей для белка или полипептида (С),включающего аминокислотную последовательность для каждого члена данной серии, или белка или полипептида (D), включающего аминокислотную последовательность, гомологическую аминокислотной последовательности каждого члена этой серии; 3) выявление локального линейного сходства (подобия) аминокислотных последовательностей у указанного белка или полипептида (А) и у отобранного белка или полипептида (С) или у установленного белка или полипептида (D); и 4) прогнозирование, является ли установленный белок или полипептид (С) и/или белок или полипептид (D) тем белком или полипептидом (В), который взаимодействует с белком или полипептидом (А), на основе результатов выявления локального линейного сходства аминокислотных последовательностей и на значении,вычисленном по частоте повторения аминокислоты и/или по частоте повторения указанных олигопептидов в вышеупомянутой базе данных по аминокислотным последовательностям. Одним воплощением настоящего изобретения может явиться вышеобозначенный способ прогнозирования, в котором олигопептид имеет длину 4-15 аминокислотных остатков. Помимо этого, в другом своем воплощении настоящее изобретение относится к среде записи, несущей программу для прогнозирования белка или полипептида (В), который взаимодействует с данным белком или полипептидом (А), которая (среда) включает в себя, по крайней мере, следующие средства от а) до f):a) средство для ввода информации об аминокислотной последовательности белка или полипептида (А) и для хранения информации;b) средство для перегруппировывания вышеуказанной информации в серию олигопептидов, имеющих заданную длину в качестве информации о последовательности, и средство для хранения полученной соответственно информации о последовательностях;c) средство для хранения введенной базы данных по белкам;d) средство для доступа к хранимой базе данных по белкам и для установления белка или полипептида (С), имеющего аминокислотную последовательность такую же, как у указанного олигопептида, или белка или полипептида (D),имеющего аминокислотную последователь 3 ность, гомологическую аминокислотной последовательности указанного олигопептида, и средство для хранения и вычисления установленного результата;e) средство для выполнения полинейной локальной сверки между белком или полипептидом (А) и установленным белком или полипептидом (С) или белком или полипептидом (D) и средство для запоминания и расчитывания результата; иf) средство для получения результирующей величины частоты встречаемости (повторяемости) аминокислоты и/или повторяемости указанного олигопептида исходя из базы данных по белкам, с последующим выводом на экран индекса для прогнозирования белокбелковых взаимодействий исходя из этой результирующей величины и из результирующей величины вышеуказанной локальной сверки, и средство для запоминания и вывода на дисплей полученного результата и для последующего установления белка или полипептида (В), который взаимодействует с белком или полипептидом (А). В следующем воплощении настоящее изобретение относится к носителю записи, включающему, по крайней мере, одно из нижеперечисляемых средств от g) до l) в дополнение к средствам от а) до f):g) средство для ранжирования силы белокбелковых взаимодействий у установленных белков или полипептидов (В), основанного на индексах, вычесленных по результирующей величине локальной сверки и по результирующей величине повторяемости аминокислоты и/или повторяемости олигопептида в белковой базе данных для случая, когда существует более одного белка или полипептида (В), который установлен, и средство для хранения и вывода результата;h) средство для вывода на дисплей полнодлиннотных аминокислотных последовательностей белка или полипептида (А) и установленного белка или полипептида (В), с последующим указанием местоположения (локализации) в полнодлиннотной последовательности частичной последовательности, которая подлежит полинейной сверке в случае, когда частичные аминокислотные последовательности сверяются путем локальной полинейной сверки между белком или полипептидом (А) и белком или полипептидом (В);i) средство для расчета пространственной структурной модели в случае, когда известна пространственная структура белка или полипептида (А) или установленного белка или полипептида (В), или в случае, когда можно построить пространственную структурную модель путем гомологического моделирования, с последующим выводом на дисплей структуры частичных аминокислотных последовательностей,которые подлежат сверке путем полинейной 4 локальной сверки между белком или полипептидом (А) и белком или полипептидом (В) на пространственной структуре;j) средство для классификации белков в базе данных по белкам для того, чтобы сузить область поиска, и для запоминания этого;k) средство для посерийного ввода каждого белка в качестве белка или полипептида (А) в базу данных по белкам; иl) средство для хранения геномной базы данных. Еще в одном своем воплощении настоящее изобретение относится к устройству для прогнозирования белок-белковых взаимодействий,включающему в состав средства, несомые вышеприведенной средой для записи. В дополнительном варианте воплощения настоящее изобретение относится к способу для спецификации белков или полипептидов, взаимодействующих друг с другом, который состоит из идентификации белка или полипептида (В),взаимодействующего согласно прогнозу, сделанному вышеприводимым прогноз-методом или прогноз-устройством, с данным конкретным белком или полипептидом (А), а затем из экспериментального подтверждения наличия взаимодействия между белком или полипептидом (А) и белком или полипептидом (В). Кроме того, в другом воплощении настоящее изобретение относится к белку или полипептиду, специфицированному вышеуказанным методом. Еще в одном воплощении настоящее изобретение относится к способу скрининга (отбора) соединения, обладающего способностью контролировать взаимодействие данного конкретного белка или полипептида (А) с белком или полипептидом (В), с использованием вышеуказанного способа прогнозирования или прогноз-устройства. И еще в одном воплощении настоящее изобретение относится к новому соединению,полученному способом скрининга, и новому соединению, способному контролировать взаимодействие белка или полипептида (А) с белком или полипептидом (В), полученным фармакоконструированием (drug-design) на основе полученной информации об этом соединении. В другом воплощении настоящее изобретение относится к олигопептиду, содержащему аминокислотную последовательность SEQ IDNo: 1, который обладает способностью контролировать взаимодействие веротоксина 2 (VTII) сBcl-2, или к олигопептиду, содержащему аминокислотную последовательность, гомологическую этому олигопептиду, и способному контролировать взаимодействие VTII с Всl-2, или к полипептиду, включающему в свой состав какой-либо из этих олигопептидов и способному контролировать взаимодействие VTII с Всl-2. В дополнение к этому еще в одном своем воплощении настоящее изобретение относится к 5 агенту против гибели клеток, содержащему олигопептид, включающий аминокислотную последовательность SEQ ID No: 1. Еще в одном воплощении настоящее изобретение относится к способу скрининга соединения, способного контролировать взаимодействие VTII с Всl-2, в котором применяются вышеупоминаемый олигопептид и/или вышеупоминаемый полипептид. И еще в одном воплощении настоящее изобретение относится к способу определения последовательности олигонуклеотида, кодирующей олигопептид, вовлекаемый во взаимодействие данного конкретного белка или полипептида (А) с белком или полипептидом (В),который согласно сделанному прогнозу взаимодействует с белком или полипептидом (А), в котором используется вышеупомянутый прогнозный способ или вышеупомянутое прогнозирующее устройство. В следующем воплощении настоящее изобретение относится к серии комбинаций человеческих белков, которые в соответствии со сделанным прогнозом взаимодействуют друг с другом и идентифицированы с помощью вышеупомянутого прогнозного способа или вышеупомянутого прогнозного устройства. Кроме того, еще в одном воплощении настоящее изобретение относится к способу селективного отбора комбинаций белков, обладающих белок-белковым взаимодействием, связанным с заболеванием; каковой метод включает отбор этой комбинации на основе информации о каком-либо известном белке, связанном с данным заболеванием, из числа вышеупомянутых серий комбинации белков. Далее, еще в одном воплощении настоящее изобретение относится к серии комбинаций белков, обладающих белок-белковым взаимодействием, связанным с заболеваниями, и которые получены вышеуказанным методом. Еще в одном воплощении настоящее изобретение относится к способу скрининга соединения, которое способно контролировать взаимодействие конкретной комбинации и/или двух белков, отобранных далее из серии комбинаций белков, имеющих белок-белковое взаимодействие, связанное с заболеваниями, полученных так, как указано выше. Еще в следующем воплощении настоящее изобретение относится к соединению, идентифицированному по способу скрининг-отбора в качестве соединения, контролирующего это взаимодействие. Еще в одном воплощении настоящее изобретение относится к способу прогнозирования процессирующего участка белка путем прогнозирования межбелкового взаимодействия конкретного белка с ферментом, расщепляющим указанный белок, с использованием вышеупомянутого способа прогнозирования или прогноз-устройства. 6 Помимо этого, в одном своем воплощении настоящее изобретение относится к аминокислотной последовательности, содержащей белокпроцессирующий участок, которая получена вышеуказанным способом для белокпроцессирующего участка, и/или к аминокислотной последовательности, содержащей частичную последовательность, гомологическую процессирующему участку. Краткое описание рисунков На фиг. 1 а показаны 20 аминокислотных остатков из аминотерминального конца веротоксина 2. На фиг. 1b показаны олигопептиды,имеющие каждый аминокислотную последовательность длиною в 5 остатков, которые были получены перегруппировкой аминокислотной последовательности, содержавшей вышеуказанные 20 остатков, в качестве информации о последовательности, с использованием программы. На фиг. 1 с показаны олигопептиды,имеющие каждый аминокислотную последовательность длиною в 6 остатков, которые были получены перегруппировкой аминокислотной последовательности, содержавшей вышеуказанные 20 остатков, в качестве информации о последовательности, с использованием программы. На фиг. 2 показаны олигопептиды, имеющие каждый аминокислотную последовательность длиною в 5 остатков, которые были получены перегруппировкой 13 остатков из аминотерминального конца веротоксина 2 в качестве информации о последовательности, с использованием программы, а также человеческие белки,содержащие аминокислотную последовательность этих олигопептидов. На фиг. 3 показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие в себя часть веротоксина 2 (VTII) и человеческой адренэнергик-рецептор-киназы 2 (ARK2). На фиг. 4 а показанаповторяемость каждой из аминокислот в белковом синтезе бактерииEscherichia coli. На фиг. 4b показано процентное содержание каждой аминокислоты в белковом синтезе бактерии Escherichia coli. Фиг. 5 показывает упрощенную последовательность средств. На фиг. 6 показаны аминокислотные последовательности олигопептидов, производных от веротоксина 2, которые присутствуют также в человеческих белках, связанных с гибелью клеток, и соответствующие человеческие белки. На фиг. 7 показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие часть веротоксина 2 (VTII) и Всl-2. На фиг. 8 показан результат локальной полинейной сверки, в результате которой были 7 получены олигопептиды, включающие часть веротоксина 2 (VTII) и Bcl-xL. На фиг. 9 показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие часть веротоксина 2 (VTII) и MCL-1. На фиг. 10 а-b показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие часть веротоксина 1 (VTI) и Всl-2 (фиг. 10 а) или BclxL (фиг. 10b). На фиг. 11a-b показаны образцы электрофореза, отражающие результат подтверждающих экспериментов с использованием клетокHepG2 и клеток В 10, которые показали, что веротоксин 2 (VTII) и Всl-2 взаимодействуют друг с другом фиг. 11 а) и (фиг. 11b) справа), но веротоксин 1 (VTI) и Bcl-2 друг с другом не взаимодействуют фиг. 11b) слева). На этих фигурах Bcl-2 IPs и VTII IPs указывают, что Bcl-2 иVTII были иммуноосаждены соответственно анти-Bcl-2 антителами и анти-VTII антителами. На фиг. 11a (слева) показан результат вестернблотта с применением анти-Bcl-2 антитела (Bcl2 WB). На фиг. 11 а показан результат вестернблотта с применением анти-VTD антитела (VTIIWB). На фиг. 11b показаны образцы электрофореза, отражающие результат подтверждения субклеточной фракции клеток В 10, которые были подвергнуты воздействию веротоксином 1(VTI) (слева) или веротоксином 2 (VTII) (справа), где эти белки были определены посредством соответственно анти-VTI антитела и антиVTII антитела. На фиг. 12 показаны участки, соответствующие локальной полинейной сверке веротоксина 2 (VTII) и Bcl-2. На фиг. 13 на пространственной структуреBcl-xL с использованием проволочной модели показана та часть, которая является гомологичной по отношению к частичной последовательности веротоксина 2 (VТII). На фиг. 14 показана, с использованием проволочной модели, та часть, которая является гомологичной по отношению к частичной аминокислотной последовательности Bcl-xL, на пространственной структуре веротоксина 2, построенной путем гомологического моделирования. На фиг. 15 показано, что олигопептидNWGRI, который включает часть веротоксина 2NWGRI. На фиг. 16 а-b показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, которые включают часть белка оболочки Т-хелперных клеток человека CD4 и поверхностного белка gp120 вируса 8 области, имеющей высокую локальную гомологичность у CD4 и gp120. На фиг. 17 показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие частьCED-4 (связанный с гибелью клетки белок нематоды) и белок МАС-1 (который связывает сCED-4). На фиг. 18 показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие часть предшественника амилоидного белка (АРР) иBASE (фермент, который расщепляет данный протеин). На фиг. 19 а-b показан результат локальной полинейной сверки, в результате которой были получены олигопептиды, включающие часть предшественника белка фурина (furin-pre) и предшественника белка фактора фон Виллебрандт (VWF-pre). На фиг. 19 а показаны олигопептиды, которые содержат часть обоих белков. На фиг. 19b показана аминокислотная последовательность той части, которая имеет высокую локальную гомологичность в обоих белках. На фиг. 20 показан результат локальной полинейной сверки, посредством которой были получены олигопептиды, включающие часть предшественника амилоидного белка (АРР) и белка РС 7 (которая, как считается, принимает участие в ходе его процессинга-созревания). Символом "=" на рисунке помечен участок, являющийся согласно прогнозу участком расщепления. Краткое описание буквенной нумерации ссылок а - Средство для вводаb - Средство для перегруппировки в серию олигопептидов и для запоминания этого с - Средство для храненияd - Средство для поиска и запоминания е - Средство для выполнения локальной полинейной сверки-выровненности и для храненияf - Средство подсчета повторяемости/вывода на дисплей из памятиg - Средство ранжирования/вывода на дисплей из памятиi - Средство вычисления пространственной структуры/память-вывода на дисплейj - Средство для запоминания белков и запоминанияk - Средство ввода по последовательностямn - Средство контроля о - Средство вывода Подробное описание изобретения Ниже приводится более подробное описание воплощений настоящего изобретения, а также принципа и способа настоящего изобретения, среды для записи, несущей программу реализации способа, функционирующего устройства, белков и полипептидов, полученных с 9 помощью данного способа и устройства. Приводимое ниже описание дается только для иллюстрации и не рассматривается как ограничивающее данное изобретение. Используемые при детальном описании технические и научные термины употребляются, как правило, в значениях, известных обычному специалисту той области, к которой имеет отношение настоящее изобретение, в противном случае, даются точные определения. Здесь же делаются отсылки к различным методологиям,известным обычному специалисту данной области. Сюда также включены в виде полных(библиографических) данных публикации и другие материалы, в которых изложены эти известные методологии и на которые делаются ссылки. В одном своем воплощении настоящее изобретение относится к способу прогнозирования межбелковых взаимодействий, которое основываются на следующей идее: белок образуется последовательностью до 20 видов аминокислот, однако, размещение этих аминокислот не является произвольным. Поэтому считается,что олигопептид, являющийся частичной последовательностью белка, играет определенную роль в каждом виде живого организма. Например, считается, что олигопептид, являющийся частью какого-либо конкретного фермента, играет роль в узнавании субстрата. Считается также, что в белке наличествует олигопептид, который играет важную роль в его взаимодействии с другими белками. Таким образом, функцию или взаимодействие белка следует рассматривать на олигопептидном уровне. Помимо того, что касается частоты встречаемости олигопептидов, частотность конкретных олигопептидов во всех белках, закодированных геномом одного организма, не является одинаковой. Некоторые олигопептиды встречаются часто в различных белках, другие - лишь изредка. Похоже, что олигопептид с низкой повторяемостью - это такой олигопептид, который является единственным в своем роде для каждого белка. Такой олигопептид, вероятно, определяет свойства или функции белка. С другой стороны, тот факт, что белки взаимодействуют друг с другом, означает, что взаимодействующие белки осуществляют функцию в кооперации друг с другом, посредством чего организм и осуществляет свою жизненную(biotical), биотическую активность. Если принять, что один олигопептид соответствует одной функции, то тогда два белка, взаимодействующих друг с другом, имеют, очевидно, один и тот же олигопептид или гомологичные олигопептиды. Кроме того, эти два белка, очевидно, имеют гомологическую структуру последовательностей в (их) иной, чем олигопептид, являющийся одинаковым (для обоих), части. В качестве одной из методик поиска подобия при анализе гомологичности двух белков известен метод сравнения путем линейной свер 004002 10 ки первичных структур (Minoru Kanahisa "Introduction to genome informatics (in Japanese)" Kyoritsu Publishing Co., Ltd., 93-104, 1996). Эта линейная сверка последовательностей включает общую линейную сверку и локальную линейную сверку. Общая сверка включает линейную сверку полных последовательностей, а локальная сверка включает местную линейную сверку только гомологических частей, извлекаемых из их последовательностей. При любом виде линейной сверки она выполняется таким образом, чтобы отношения между/среди двух или более последовательностей могли быть как можно более наглядными. Существующее множество комбинаций в линейной сверке находится в зависимости от длины последовательностей. В число методов выполнения комбинаторной оптимизации входит метод динамического программирования, метод СмитВатермана (Smith, T.F. and Waterman, M.S., J.Mol. Biol. 147, 195-197, 1981), основанный на том принципе, что динамическое программирование обеспечивает функциональную оценку при комбинаторной оптимизации последовательностей. Величина функции оценки, т.е. показатель гомологичности в баллах, или просто бальный показатель позволяет оценивать гомологичность двух белков. Можно приблизительно подсчитать, что чем выше бальный показатель у сравниваемых белков, тем выше гомологичность у этих белков.Что касается локальной линейной сверки, то она выполняется установлением порогового значения счета баллов,после чего выполняется комбинаторная оптимизация частичных последовательностей, при которой комбинация последовательностей отыскивается путем динамического программирования (e.g., Gotoh, О., Pattern matching of biological sequences with limited storage, Comput. Appl.Biosci. 3, 17-20, 1987). Метод локальной линейной сверки допускает поиск гомологической структуры в части белка, иной, чем та, где расположен олигопептид, являющийся одинаковой частью у двух белков. Вероятно, межбелковые взаимодействия сохранились в процессе эволюции. Пример веротоксина бактерии Escherichia coli и Bcl-2, как это описано далее, показывает, что одна функция сохраняется в белок-белковом взаимодействии вне зависимости от видов, отсюда, может быть, и структурноподобные последовательности аминокислот. Кроме того, при процессировании предшественника амилоидного белка и предшественника и белка фактора фон Виллебранд Фактор (VWF), как это описывается далее,функция протеолиза, может быть, сохранялась при взаимодействии белок-белок, и структурноподобные последовательности аминокислот,возможно, существуют. Изобретенный способ прогнозирования межбелковых взаимодействий, основанный на вышеприведенной идее, может сделать возмож 11 ным также прогнозирование сети функций, известной ранее в виде одиночной функции, а также может дать новое представление о жизни на основе результатов, которые были спрогнозированы на компьютере, и на основе всеобщей связи явлений, и оно будет весьма отличаться от того представления о жизни, что сложилось в ходе аккумулирования фактов, получаемых на основе принципа перечисления в молекулярной биологии. Кроме того, если прогнозирование взаимодействий возможно не только в одном организме, но и между двумя организмами, например между людьми и патогенными микроорганизмами, то тогда, очевидно, станет возможным объяснить патогенез, до сих пор не нашедший объяснения. Одно конкретное воплощение вышеуказанного способа прогнозирования межбелковых взаимодействий представляет собой способ извлечения и прогнозирования контрбелка из базы данных по белкам и т.п., который взаимодействует с белком, получаемым путем геномного анализа или cDNA-анализа, и функция которого неизвестна, или с белком, чья функция известна; который включает, например, следующие 1-4 стадии. На этапе 1 последовательность аминокислот конкретного белка или полипептида перегруппировывается в серию олигопептидов,имеющих заданную длину в качестве информации о последовательности. На этапе 2 определяются белки или полипептиды, которые содержат каждый из олигопептидов. На этапе 3 делается приблизительная оценка гомологичности частичных структур белков путем локальной линейной сверки. На этапе 4 делается дальнейшая оценка каждого олигопептида по частоте встречаемости. Каждый этап будет описан ниже более детально. Этап 1. Аминокислотная последовательность конкретного белка или полипептида (А), как, например, белка или полипептида, полученного геномным анализом или cDNA-анализом и чья функция неизвестна, или белка или полипептида с известной функцией, преобразовывается путем декомпозиции-перегруппировки в олигопептиды как информацию о последовательности путем шифтинга - посерийно, начиная с одного аминокислотного остатка из аминотерминального конца и до карбоксильного конца. Например, на фиг. 1 а-b показаны олигопептиды (фиг. 1b), имеющие длину из 5 аминокислотных остатков, которые были получены перегруппировкой первых 20 остатков (фиг. 1 а) веротоксина 2(VTII) бактерии Escherichia coli 0157: Н-7 из его аминотерминального конца в виде информации о последовательностях. Аминокислоты и олигопептиды далее изображаются их однобуквенными символами. 12 При выполнении этапа 1 длина олигопептида, перегруппированного в виде информации о последовательности, составляет от 4 до 15 аминокислотных остатков, предпочтительно от 4 до 8 остатков. Чем больше длина олигопептида, тем выше специфичность олигопептида, как это показано в примере 2. Этап 2. На этапе 2 осуществляется поиск белка или полипептида (С), включающего аминокислотную последовательность олигопептида, полученного путем перегруппировки на этапе 1,или белка или полипептида (D), имеющего аминокислотную последовательность, гомологическую последовательности олигопептида, среди аминокислотных последовательностей базы данных по белкам или полипептидам. Количество установленных белков или полипептидов(С) или (D) может оказаться большим или оно может оказаться зависящим от того, какой олигопептид используется. Например, на фиг. 2 показаны результаты поиска - в базе данных по белкам (SWISS-PROT версия 35) - белков, имеющих 9 олигопептидов,содержащих каждый по 5 аминокислот, полученных перегруппировкой 13 остатков веротоксина 2 (VTII) бактерии Escherichia coli 0157:H7 из аминотерминального конца. Веротоксин 2 вызывает пищевые отравления и/или поражение почек у человека, так что человеческий белок может быть мишенью при поиске контр(обратных)-белков, которые взаимодействовали бы с VTII. Например, подобный поиск показывает, что человеческий белок, включающий в себя олигопептид KCILF, изображенный на фиг. 2 (второй), - это -адренергический рецептор киназы 2 (ARK2 HUMAN). Этап 3. Производится локальная линейная сверка вышеупомянутого белка или полипептида (А) и белка или полипептида (С) или белка или полипептида (D), полученного в результате поиска на этапе 2. Например, фиг. 3 иллюстрирует результат локальной сверки веротоксина 2 (VTII) и адренергического рецептора киназы 2 (ARK2). Этап 4. Если результат локальной сверки на этапе 3 отражает в той или иной степени гомологичность частичной последовательности у вышеупоминаемого белка или полипептида (А) и у установленного белка или полипептида (С) и/или у белка или полипептида (D), то в этом случае делается прогноз, что белок или полипептид (С) и/или белок или полипептид (D) являются/является, вероятно, белком или полипептидом (В), взаимодействующим с белком или полипептидом (А). Более того, повторяемость аминокислоты/кислот и/или частотность олигопептида, который присутствует как в белке или полипептиде (А), так и в установленном 13 белке или полипептиде (С) и/или в белке или полипептиде (D), подсчитывается по базе данных по белкам; этому сопутствует общая оценка специфичности каждого из олигопептидов в базе данных по белкам или в геноме организма,содержащем белок или полипептид (А) или установленный белок или полипептид (С) и/или белок или полипептид (D). Если специфичность оказывается высокой, то и надежность прогнозирования того, что белок или полипептид (С) и/или белок или полипептид (D) являются/является белком или полипептидом (В), который взаимодействует с белком или полипептидом (А), оценивается как высокая. Например, подсчитано, что индекс специфичности вышеупомянутого олигопептидаKCILF составляет, исходя из композиционной пропорции, 1284,86 х 10-10 (фиг. 4b), которая просчитывается по повторяемости аминокислоты(фиг. 4 а) во всех белках, кодируемых геномом бактерии Escherichia coli; он показан на фиг. 4 так что специфичность эта высока. Состоящий из 5 аминокислот олигопептид, имеющий по подсчетам низкую специфичность в геноме бактерии E.coli, - это LLLLL, т.е. его индекс специфичности составляет 136344,34 х 10-10. Имеющий по подсчетам наивысшую специфичность олигопептид с 5 аминокислотами в составе - это ССССС с индексом специфичности 2,208 х 10-10,однако, этот олигопептид не встречается в геноме E.coli. Таким образом, прогноз относительно того, что веротоксин 2 будет взаимодействовать с -адренергическим рецептором киназы 2, расценивается как имеющий высокую надежность исходя из значения индекса специфичности. Для дальнейшего подтверждения белокбелкового взаимодействия можно клонировать ген, кодирующий этот белок, для экспрессии на основе информации о полученных белках, взаимодействующих друг с другом. Например, как это будет показано далее в примерах, с помощью вышеописанного способа или с помощью устройства для прогнозирования, несущего программу по способу прогнозирования, было спрогнозировано, что VTII и Bcl-2 взаимодействуют друг с другом. Это могло быть подтверждено экспериментами, в которых клетки, в которых экспрессировался Bcl-2, и клетки, в которых Bcl-2 не экспрессировался, подвергались обработке VTII, после чего следовало соиммуноосаждение с помощью антитела антиВсl-2 и антитела анти-VTII. Помимо этого, также возможно определить этот олигопептид как важный участок взаимодействия путем, к примеру, введения каким-либо из хорошо известных способов одной из мутаций в аминокислотную последовательность того олигопептида,который в соответствии со сделанным прогнозом является участком взаимодействия, и далее убедиться, что взаимодействие утрачено. Методы экспериментального подтверждения взаимо 004002 14 действия не ограничиваются вышеприведенными, любая из подходящих методик может быть использована специалистами для этой цели. Помимо этого, если будет подтверждено,что олигопептид, представляющий собой в соответствии с прогнозом участок взаимодействия, прерывает белок-белковое взаимодействие и подавляет любую функцию или акцию данного белка, то в этом случае такой олигопептид можно целенаправленно использовать в качестве фактора подавления действия данного белка. Например, как это описано в одном из нижеследующих примеров, олигопептид NWGRI, являющийся согласно сделанному прогнозу участком взаимодействия с VTII и с Bcl-2, подавляет гибель клеток, вызываемую VTII, и его можно применить в качестве фактора, направленного против гибели клеток. Подобные вещества с низким молекулярным весом можно применять в качестве фармацевтических препаратов, реагентов и т.п. Теперь будет дано описание носителя записи и устройства, содержащее программу для вышеозначенного метода прогнозирования белок-белкового взаимодействия. Упоминаемые носитель записи и устройство включают, как минимум, следующие средства от (а) до (f). На фиг. 5 приводится пример их возможной общей структуры. Ввод средств (а): Средства для ввода информации о последовательности аминокислот в конкретном белке или полипептиде (А), типа белка или полипептида, полученного путем геномного анализа илиcDNA-анализа, функция которого неизвестна,или белка или полипептида, чья функция известна. Средство перегруппировки в серию олигопептидов и для запоминания этого (b): Средство для перегруппировки информации о последовательности аминокислот, которая была введена с помощью средства ввода (а), в серию олигопептидов, обладающих заданной длиной в качестве информации о последовательности, путем шифтинга посерийно, начиная с аминокислотного остатка из аминотерминального конца до карбоксильного конца, и для запоминания полученного результата. Средство хранения (с): Средство для хранения базы данных, введенной в связи с белком и полипептидом. Средство поиска/хранения (d): Средство для доступа к базе данных по белку и полипептиду, хранимой с помощью средства хранения (с), за чем следует поиск белка или полипептида (С), включающее аминокислотную последовательность вышеупомянутого олигопептида, или белка, или полипептида(D), содержащего аминокислотную последовательность, гомологическую аминокислотной последовательности вышеуказанного олигопеп 15 тида, и (средство) для хранения полученного результата. Средство выполнения локальной линейной сверки/хранения (е): Средство для выполнения локальной сверки между вышеупомянутым белком или полипептидом (А) и отобранным белком или полипептидом (С) и/или (D), и (средство) для хранения полученного результата. Средство подсчета частотности (повторяемости)/запоминания-демонстрации (f): Средство для вычисления индекса для прогнозирования белок-белковых взаимодействий по результату вышеупомянутой локальной сверки и по результату, полученному вычислением частотности аминокислоты и/или частотности олигопептида в базе данных по белкам и полипептидам, и (средство) для хранения и демонстрации результата. В дополнение к вышеперечисленному приводимая схема для прогнозирования белокбелкового взаимодействия может быть дополнена соответствующим сочетанием следующих средств от (g) до (l). Средство ранжирования/запоминаниядемонстрации (g): Средство, предназначенное для ранжирования белков или полипептидов (В), для случаев, когда отобрано более одного белка или полипептида (В), с использованием результата локальной сверки и результата подсчета частотности аминокислоты и/или частотности олигопептида по белковой базе данных в качестве индексов, а также предназначенное для хранения/демонстрации результата. Средство локационной индикации (h): Средство для демонстрации полной длины последовательности аминокислот белка или полипептида (А) и белка или полипептида (В),сопровождаемое индикаторным указанием на месторасположение частичной последовательности, которую нужно сверить с полнодлиннотными последовательностями для случаев, когда проводят сверку частичных аминокислотных последовательностей у белка или полипептида(А) и у отобранного белка или полипептида (В). Средство просчитывания стереоструктуры/запоминания-демонстрации (i): Средство для вычисления пространственной структурной модели и последующей демонстрации структуры частичных последовательностей аминокислот, сверяемых у белка или полипептида (А) и у белка или полипептида (В) по пространственной структуре, для случаев, когда известна объемная структура белка или полипептида (А) или отобранного белка или полипептида (В), или для случаев, когда пространственная структурная модель может быть построена путем гомологического моделирования. Средство классифицирования белков/хранения (j): 16 Средство, предназначенное для классифицирования белков или полипептидов в белковой или полипептидной базе данных по их свойствам и/или по происхождению, для того, чтобы сузить область поиска, и далее для их хранения. Средство последовательного ввода (k): Средство для последовательного ввода каждого белка или полипептида в качестве белка или полипептида (А) в базу данных по белкам или полипептидам. Средство хранения (l): Средство, предназначенное для хранения геномной базы данных. Вышеперечисленные средства функционируют на соответствующем носителе. В качестве одного из воплощений изобретения на практике для каждого из этих средств можно предусмотреть устройство, содержащее носитель записи, избирательно включая его в виде программы. Устройство для прогнозирования белок-белковых взаимодействий действует так, как это описано ниже (см. фиг. 5). Конкретный белок или полипептид (А),такой как белок или пептид, введенный с помощью средства (а), и функция которого может быть известна или неизвестна, перегруппировывается с помощью средства перегруппировки и запоминания (b) в серию олигопептидов, имеющих заданную длину, в качестве последовательностной информации, и эти олигопептиды запоминаются. В данном случае белок или полипептид (А) вводится, по желанию, последовательно с помощью средства последовательного ввода (k) из средства (с), которое хранит базу данных, введенную в связи с данным белком или полипептидом. Поиск аминокислотной последовательности запомненного выше олигопептида осуществляется через средство (с) с помощью средства поиска/хранения (d) и далее отбирается и запоминается белок или полипептид (С), содержащий аминокислотную последовательность этого олигопептида, или белок и полипептид (D), содержащий аминокислотную последовательность, гомологическую аминокислотной последовательности этого олигопептида. При поиске также можно классифицировать белки или полипептиды в базе данных для того,чтобы сузить область поиска, с помощью средства классификации белков/хранения (j), после чего проводится поиск в результирующей области. Указанный белок или полипептид (А) и отобранный белок или полипептид (С) или (D) подвергаются локальной линейной сверке с помощью средства локальной сверки/запоминания-демонстрации (е), полученный результат запоминается. Затем с помощью средства подсчета частотности/хранения (f) подсчитывается частота встречаемости аминокислоты и/или частота встречаемости олигопептида по базе данных, хранимой средством (с), и на основании этого результата и полученного и запомненного ранее результата локальной сверки вычисляется 17 индекс для прогнозирования белок-белковых взаимодействий. Затем то, что получено, отображается на дисплее устройства, а именно то,что является белком или полипептидом (С) или(D), которые в соответствии с прогнозом могут взаимодействовать с вышеуказанным белком или полипептидом (А); аминокислотная последовательность олигопептида, являющегося общим для этих белков; частотность этого олигопептида; индексы для прогнозирования белокбелковых взаимодействий и т.п. Выведенные на дисплее результаты позволяют определить по индексам для прогнозирования белок-белковых взаимодействий, который из белков или полипептидов является белком или полипептидом(В), взаимодействующим с вышеуказанным белком или полипептидом (А). Кроме того, в связи с вышеупоминаемым белком или полипептидом (В) можно также разместить на дисплее функциональную информацию о белке,которая сохраняется в средстве (с), а также генную информацию из средства (l), заводимую по желанию, в которой хранится геномная база данных. Когда отобранными оказываются более одного белка или полипептида (В), средство ранжирования/запоминания-демонстрации (g) позволяет сделать ранжирование этого белка или полипептида (В) по степени специфичности для взаимодействия с данным белком или полипептидом (А). Можно также использовать средство локационной индикации (h) для индикаторного указания, которая из частей полнодлиннотных аминокислотных последовательностей данного белка или полипептида (А) и установленного белка или полипептида (В) является той частичной последовательностью аминокислот, что подлежит линейной сверке у белка или полипептида (А) и у белка или полипептида (В). Помимо этого, можно также вывести на дисплей пространственную структуру белка или полипептида (А) и белка или полипептида (В), равно как и ту их часть, что подлежит сверке у белка или полипептида (А) и у белка или полипептида(В), с помощью средства просчитывания стереоструктуры/запоминания-демонстрации (i). Устройство может быть снабжено клавиатурой (m),средством контроля (n) и средством вывода (о),которые тоже отображены на фиг. 5, и т.п., равно как и все эти средства (а)-(l). Данный способ прогнозирования белокбелковых взаимодействий или данное прогнозустройство могут быть также использованы для отсортировывания нового соединения, которое способно контролировать взаимодействие белка или полипептида (A) с белком или полипептидом (В). Этот метод отсортировки нового соединения, которое способно контролировать взаимодействие данного белка или полипептида(А) с белком или полипептидом (В), выполняется на основе информации о последовательности аминокислот ключевого олигопептида. Аминокислотная последовательность отобранного 18 олигопептида, аминокислотная последовательность олигопептида, гомологическая первой,или полипептид, включающий эту аминокислотную последовательность, или гомологическая последовательность аминокислот per se могут оказаться способными контролировать взаимодействие белка или полипептида (А) и белка или полипептида (В). Например, если существует белок или полипептид (В), обладающий функцией рецептора по отношению к белку или полипептиду (А), то вполне вероятно, что олигопептид, который будет отсортирован вышеописанным способом, явится антагонистом по отношению к взаимодействию белка или полипептида (А) с белком или полипептидом (В). Например, в случае, когда белок или полипептид (А) активирован взаимодействием с белком или полипептидом (B), вполне вероятно, что олигопептид, отсортированный вышеуказанным способом, обладает действием антагониста. В реальности, как это описывается подробно в примерах ниже, экспериментально было подтверждено, что олигопептид NWGRI, описанный в SEQ ID NO: 1, содержащий порциюVTII и Bcl-2, в отношении которых было спрогнозировано с помощью настоящего изобретения и экспериментально подтверждено, что они взаимодействуют друг с другом, прерывает формирование комплекса в результате взаимодействия VTII с Bcl-2 и подавляет гибель клеток, индуцируемую VTII. Следовательно, олигопептид NWGRI может использоваться в качестве лекарственного средства для контролирования заболевания, связанного с гибелью клеток, индуцированной VTII, например в качестве лекарственного средства для лечения заболевания, вызываемого бактерией Escherichia coli 0157, экспрессирующей VTII, или более конкретно в качестве силы, противодействующей гибели клеток. Более того, олигопептид, имеющий аминокислотную последовательность, гомологическую этой аминокислотной последовательности, и способный контролировать взаимодействие VTII с Bcl-2, или полипептид,включающий эту аминокислотную последовательность или аминокислотную последовательность, гомологическую этой аминокислотной последовательности, и способный контролировать взаимодействие VTII с Bcl-2, также может быть использован в качестве лекарственного средства для контролирования заболевания, связанного с гибелью клеток, вызываемой VTII. Помимо этого, можно получить новое соединение, способное контролировать взаимодействиеVTII с Bcl-2, с использованием этих олигопептидов и полипептидов по методу конструирования лекарств (drug design) или с применением какого-либо из известных скрининг-методов. Таким образом, новое соединение, получаемое в результате драг-дизайна на основе информации об олигопептиде, полученном вышеупоминаемым скрининг-методом в соответст 19 вии с одним из воплощений настоящего изобретения, является способным контролировать взаимодействие данного конкретного белка или полипептида (А) с белком или полипептидом(В). А именно, прогнозирование взаимодействия названных белка или полипептида (А) и белка или полипептида (В) позволяет сделать производное олигопептида, полученного вышеприводимым скрининг-методом, а также соединение с низким молекулярным весом, имеющее структуру, гомологическую этому олигопептиду, путем хорошо известного метода драг-дизайн. Вышеприводимый способ прогнозирования является также полезным в качестве способа определения последовательности олигонуклеотида, кодирующего олигопептид, вовлекаемый во взаимодействие данного конкретного белка или полипептида (А) с белком или полипептидом (В). Применение хорошо известных методик, таких как замещение, делиция, добавление, вставка или индуцированная мутация,основанное на этой информации, дает возможность получить полезный олигонуклеотид. Полученный олигонуклеотид можно использовать для получения соединения, способного контролировать на генном уровне взаимодействие белка или полипептида (А) с белком или полипептидом (В). Например, он используется для получения антисмыслового олигонуклеотида для прерывания белок-белкового взаимодействия. Помимо того, полученный олигонуклеотид может быть использован при диагностировании заболевания, связанного с белок-белковым взаимодействием. В другом своем воплощении настоящее изобретение относится к серии комбинаций человеческих белков, в отношении которых спрогнозировано, что они имеют белокбелковое взаимодействие, что спрогнозировано с помощью вышеприводимого способа или устройства для прогнозирования белок-белковых взаимодействий. Эта серия комбинации белков может быть использована как каталог или как база данных. Можно получить серию комбинаций белков, взаимодействующих друг с другом,из тех, что вовлечены в заболевание, путем отбора таких, которые вступают в белок-белковое взаимодействие с теми, что связаны с заболеванием, на основе информации об известных белках, которые могут иметь отношение к данному заболеванию, исходя из серии комбинации белков, имеющих белок-белковое взаимодействие. Это может служить каталогом или базой данных. Эти комбинации белков полезны как лекарственные средства для лечения или профилактики заболеваний или как средство для получения лекарств. Например, соединение, обладающее способностью контролирования взаимодействия двух белков, можно получить путем отбора, используя хорошо известный скринингметод, и путем использования получаемой комбинации протеинов. 20 В случае, если среди комбинаций, состоящих из двух белков, обладающих белокбелковым взаимодействием, один белок является ферментом, способным к процессированию белка, и он расщепляет другой белок, процессирующий участок белка, который расщепляется,можно спрогнозировать вышеприводимым способом или с помощью устройства для прогнозирования белок-белкового взаимодействия. Например, как показывается в примере ниже, возможно сделать прогноз для субъекта взаимодействия предшественника амилоидного белка с ферментом, вовлеченным в этот процесс, и для субъекта взаимодействия предшественника белка фактора фон Виллебранд с ферментом фурином, вовлеченным в этот процесс. А именно, вышеприведенный способ или устройство для прогнозирования белок-белковых взаимодействий делает возможным прогнозировать участок расщепления, когда белокпредшественник расщепляется для того, чтобы взаимодействовать со зрелым белком. Таким образом, неизвестный до сих пор фермент, обладающий свойством процессировать белок,имеющий отношение к заболеванию, и белок,который расщепляется этим ферментом, могут быть получены путем прогнозирования белокбелкового взаимодействия. Примеры Хотя преимущества, особенности и возможности применения данного изобретения весьма подробно изложены ниже со ссылками на воплощения данного изобретения в качестве примеров, данное изобретение не ограничивается нижеследующими примерами. Помимо этого,несмотря на то, что в нижеследующих примерах в качестве белковой базы данных использовалась SWISS-PROT версия 35, возможно также пользоваться и другими базами данных по белкам или аналогичными им. Пример 1. На фиг. 1 а-с продемонстрированы олигопептиды, перегруппированные из первых 20 остатков (фиг. 1 а) веротоксина 2 (VTII) бактерии Escherichia coli 0157:H7 с аминоконцевого края, где олигопептиды имеют аминокислотную последовательность длиной в 5 остатков (фиг. 1b) в качестве примера этапа 1. На фиг. 1 с продемонстрированы олигопептиды, перегруппированные из первых 20 остатков веротоксина 2(VTII) с аминоконцевого края, где олигопептиды имеют аминокислотную последовательность длиной в 6 остатков. Пример 2. На этапе 4 вышеизложенного способа прогнозирования взаимодействий белок-белок числовые величины используются в качестве показателя для прогноза взаимодействия белков или полипептидов. Величины рассчитываются из частоты встречаемости аминокислоты в белковой или полипептидной базе данных и частоты встречаемости олигопептида в белковой или 21 полипептидной базе данных. В качестве примера вычисляется специфичность олигопептидов по частоте встречаемости аминокислоты во всех белках, закодированных геномом Escherichiacoli, как показано на фиг. 4 а-b. Процентное содержание 'Аi' каждой из аминокислот 'аi' может быть вычислено по частоте встречаемости (фиг. 4 а) 20 видов аминокислот во всех белках, закодированных геномом Escherichia coli. Специфичность олигопептида а 1 а 2 а 3 а 4 а 5 рассчитывается как А 1 хА 2 хА 3 хА 4 хА 5. Например,для олигопептида KCILF подсчитано, что она равна 4,406610 х 1,170608 х 6,004305 х 10,639652 х 3,898962 х 10-10. Специфичность олигопептида LLLLL составляет по расчету 136344,34 х 10-10, а специфичность олигопептида ССССС равна 2,20 х 10-10. Чем меньше числовое значение, тем выше специфичность олигопептида. Олигопептидом,обладающим самой высокой специфичностью среди всех, имеющих последовательность аминокислот длиною в 5 остатков, является олигопептид ССССС, однако, этот олигопептид не встречается ни в одном из белков, закодированных геномом Escherichia coli. Напротив, наименьшую специфичность имеет олигопептидLLLLL. Когда белок или полипептид (А) перегруппировывается в олигопептиды на этапе 1,то, чем больше длина олигопептида, тем выше специфичность этого олигопептида. Пример 3. На этапе 4 вышеизложенного способа прогнозирования взаимодействий белок-белок в качестве показателя прогноза взаимодействия белков или полипептидов используется результат локальной линейной сверки. Здесь приведен пример, в котором использованы балльные показатели сходства частичной последовательности по методу Гото (Gotoh, О., Pattern matchingAppl. Biosci. 3, 17-20, 1987). В следующих примерах, где показатель соответствия составлял 25,0 или более баллов, считалось, что неполные последовательности аминокислот сходны (гомологичны) между белком или полипептидом(А) и другим белком или полипептидом (В). Принимается, что неполные последовательности аминокислот "m" белка или полипептида (А) и другого белка или полипептида (В) сходны, если их балльные показатели равны Si(1im), а длина аминокислотной последовательности белка или полипептида (В) составляет LB. Показатель для прогнозирования взаимодействия белка или полипептида (А) с белком или полипептидом (В), вычисленный по результату локального сходства, определяется как сумма (SI)/LB. В соответствии с прогнозом, чем выше данный показатель, тем сильнее взаимодействие. Пример 4. Прогноз взаимодействия VTII с 22 Веротоксин 2 (VTII) Escherichia coli 0157:H7 вызывает у человека пищевое отравление и поражения почек, однако, механизм действия недостаточно известен (Sandvig, К., et al.,Exp. Med. Biol. 412, 225-232, 1997; Paton, J.C.,and Paton, A.W., Clin. Microbiol. Rev. 11, 450479, 1998). Данный белок является токсичным белком. Таким образом, человеческие белки,связанные с гибелью клеток, являются кандидатами в белки, взаимодействующими с данным белком. Так, в белковой базе данных SWISSPROT версия 35 был проведен поиск человеческих белков, могущих взаимодействовать с данным белком, в особенности человеческих белков, связанных с гибелью клетки, и ниже приведен соответствующий пример, демонстрирующий, что эти белки действительно взаимодействуют между собой. В SWISS-PROT версии 35 из числа олигопептидов, имеющих аминокислотную последовательность длиной в 5 остатков веротоксина 2,были найдены следующие четыре, которые содержатся в человеческом белке, относящемся к гибели клетки, а именно LCLLL, QRVAA,EFSGN и NWGRI (см. фиг. 6, на котором человеческие белки показаны с помощью идентификаторов белков согласно SWISS-PROT версии 35). Значения специфичности для данных олигопептидов были вычислены по частоте встречаемости аминокислот во всех белках, закодированных геномом Escherichia coli, и показаны на фиг. 4 а-b, то есть специфичность олигопептида LCLLL составила 15001,03 х 10-10, олигопептида QRVAA - 15584,55 х 10-10, EFSGN 3801,65 х 10-10 и NWGRI - 1479,85 х 10-10. Как было установлено, NWGRI имеет наивысшую специфичность среди этих четырех олигопептидов. Олигопептид NWGRI содержит в себе часть веротоксина 2 и каждый из трех человеческих белков, т.е. Bcl-2, Bcl-xL и MCL-1. Локальное сходство между веротоксином 2 (VTII) и каждым из Bcl-2, Bcl-xL и MCL-1 показало частичную гомологию в их аминокислотной последовательности, как показано на фиг. 7, 8 и 9. Затем сумма балльных показателей локального сходства делилась на длину каждого из белков для получения показателя, описанного в примере 3 и указанного ниже.MCL-1 (34,0+30,0+28,0+26,0)/350=0,337 Среди этих трех белков Bcl-2 и Bcl-xL образуют общую семейную группу. На основе этого показателя, вычисленного из локального сходства вышеописанным способом, сделан прогноз, что Bcl-2 и Bcl-xL обладают более сильным взаимодействием с веротоксином 2 среди белков Bcl-2, Bcl-xL и MCL-1. Веротоксин 1 (VTI) - это один из веротоксинов,вырабатываемыхcoli 0157:H7, он является изоформой веротоксина 2. Токсичность веротоксина 1 слабее, чем у веро 23 токсина 2, причем первая составляет около 1/50 последней (Tesh, V.L., et al., 1993, Infect. Immun. 61, 3392-3402). В белковой базе данных SWISSPROT версия 35 человеческий белок, содержащий олигопептид с аминокислотной длиной 5 остатков, составляющий часть веротоксина 1 и относящийся к гибели клетки, - этоP2X1HUMAN, а олигопептид - SSTLG. Однако специфичность олигопептида SSTLG, для которой вычислено значение 14385,63 х 10-10 по частоте встречаемости аминокислоты во всех белках, закодированных геномом Escherichia coli,показанное на фиг. 4 а-b, является более низкой,чем у NWGRI, приблизительно на 1/10. Для веротоксина 1 олигопептид NWGRL,который соответствует олигопептиду NWGRI с аминокислотной длиной 5 остатков в веротоксине 2,обнаруживает специфичность 2622,30 х 10-10, вычисленную по фиг. 4 а-b, что ниже, чем у NWGRI. Как Bcl-2, так и Bcl-xL содержат олигопептид NWGR с длиной аминокислотной последовательности 4 остатка, который составляет часть веротоксина 1. Сравнение величин специфичности NWGRI и NWGRL позволяет прогнозировать, что как Bcl-2, так и BclxL взаимодействуют с веротоксином 2 сильнее,нежели с веротоксином 1. Помимо этого, показатели, полученные расчетами по результату(фиг. 10) локальной выровненности между веротоксином 1 и Bcl-2 или Bcl-xL, составляют соответственно (27,0+26,0)/239=0,222 и 26,0/233=0,112(иной гомологической частичной аминокислотной последовательности, чем NWGR, нет). Отсюда прогнозируется, что взаимодействие веротоксина 1 с Bcl-2 или Bcl-xL значительно слабее, нежели взаимодействие веротоксина 2 сBcl-2 или Bcl-xL. Пример 5. Экспериментальное подтверждение прогноза взаимодействия VTII и Bcl-2. В примере 4 достоверность прогноза того,что веротоксин 2 взаимодействует с человеческими Bcl-2 или Bcl-xL, полагалась высокой. На основании результата данного прогноза было экспериментальным путем подтверждено, что веротоксин 2 действительно взаимодействует сBcl-2 (фиг. 11a и фиг. 11b (правая. В частности, раковая клетка печени человека HepG2 (не экспрессирующая в существенной степени генBcl-2) и клетки В 10, полученные путем трансдукции Bcl-2-экспрессирующего вектора вHepG2, были обработаны веротоксином 2(VTII), а затем было произведено совместное иммуноосаждение с использованием антител против-Bcl-2 (Bcl-2 IPs) и против-VTII (VTIIIPs). На фиг. 11 а (левая) демонстрируется результат анализа Western Blotting с использованием антител против-Bcl-2 после совместного иммуноосаждения; на фиг. 11b (правая) демонстриуется результат анализа Western Blotting с использованием антител против-VTII. На основании этих результатов было подтверждено, что 24 в клетках В 10 был совместно иммуноосажден комплекс VTII/Bcl-2, т.е. данные два белка взаимодействуют между собой. Кроме того,клетки В 10 обрабатывались веротоксином 1(VTI) или веротоксином 2 (VTII) с целью исследовать при помощи антител проти-VTI и против-VTII, в какой субклеточной фракции данные белки были обнаружены. Bcl-2 в митохондриях играет весьма важную роль в клеточной смерти. Веротоксин 2 (VTII) также был обнаружен в митохондриальной фракции (фиг. 11b(правая. С другой стороны, веротоксин 1 не был обнаружен в митохондриальной фракции. Таким образом, было экспериментально доказано,что веротоксин 1 не обладает сильным взаимодействием с Bcl-2 митохондрий. Данный результат показан на фиг. 11b (левая). Пример 6. На фиг. 12 иллюстрируется пример, в котором полные аминокислотные последовательности веротоксина 2 (VTII) и Bcl-2 продемонстрированы таким образом, чтобы показать в полных последовательностях местоположение сходных участков последовательностей. Пример 7. Стереоструктура Bcl-xL известна и зарегистрирована в PDB, которая является базой данных по белковым стереоструктурам. На основании результата локальной линейной сверки сходства фиг. 8 жирными линиями на фиг. 13 показаны участки аминокислотных последовательностей в стереоструктуре Bcl-xL, гомологические участкам аминокислотных последовательностей веротоксина 2. Пример 8. Считается, что веротоксин 2 расщепляет часть рибосомной РНК, таким образом останавливая синтез белка, в чем проявляется его токсичность. Стереоструктура белка рицин, расщепляющего участок рибосомной РНК, зарегистрирована в PDB, которая является базой данных белковых стереоструктур. На основе данной структуры было произведено гомологическое моделирование веротоксина 2. На основании результата локальной линейной сверки сходства фиг. 8 в стереоструктурной модели на фиг. 14 жирными линиями показаны участки аминокислотных последовательностей, гомологические участкам аминокислотных последовательностей Bcl-xL. Пример 9. Подавление олигопептидомNWGRI индуцирования клеточной смерти веротоксином 2. Далее было экспериментально подтверждено, что олигопептид NWGRI (SEQ ID NO: 1), который был обнаружен в примере 4 и составляет часть VTII и Bcl-2, может контролировать взаимодействие VTII с Bcl-2. Прежде всего,образование данного комплекса было исследовано с использованием экстракта клеток В 10,экспрессирующих Bcl-2, применявшихся в при 25 мере 5, и биотинилированного VTII в присутствии олигопептида, а затем проанализировано путем Far Western blotting analysis. ОлигопептидNWGRI прерывал образование комплекса VTII и Bcl-2 в степени, зависящей от дозировки. Кроме этого, клетки В 10 были предварительно обработаны олигопептидом NWGRI при 0, 10, 50, 100 мкМ, а затем обрабатывались VTII по 10 нг/мл в течение 24 ч, при этом производился количественный анализ индукции клеточной смерти апоптозом. В соответствии с общепринятой методикой с помощью Hoechst 33342/PI (Propidium iodide) было окрашено всего около 5000 ядер; соотношение ядер, демонстрирующих апоптоз, показано на фиг. 15. Как показывает рисунок, приблизительно 85% клеток вызвали апоптотическую клеточную смерть при обработке только веротоксином 2 (VTII), в то время как при предварительной обработке олигопептидом NWGRI индукция апоптотической клеточной смерти подавлялась в степени,находящейся в зависимости от дозировки. Таким образом, было подтверждено, что олигопептид NWGRI, составляющий часть VTII иBcl-2, прерывает взаимодействие VTD с Bcl-2 таким образом, что ингибирует образование комплекса данных белков и, вследствие этого,подавляет индукцию клеточной смерти VTII. Пример 10. CD4/gp120HIV-l. Человеческий вирус СПИДа HIV-1 инфицирует хелпервые Т-клетки. Важным первым этапом инфицирования данных клеток является связывание белка gp120 на поверхности вируса с поверхностным белком CD4 хелперных Тклеток. В данном примере было изучено, возможно ли спрогнозировать связывание gp120 иCD4 с помощью вышеизложенного способа прогнозирования. Протеин CD4 был разложен на олигопептиды с длиной аминокислотной последовательности в 5 остатков, а в белковой базе данных был проведен последовательный поиск белков,имеющих аминокислотную последовательность олигопептида, производного от CD4, при этом был выделен gp120 как белок, содержащий олигопептид SLWDQ (фиг. 16 а). ОлигопептидSLWDQ существует только в белке CD4 из человеческих белков по SWISS-PROT версии 35,т.е. его частота встречаемости в человеческих белках равна 1, а специфичность очень высока. Кроме того, помимо этого олигопептида, существует локально-гомологическая область (фиг. 16b). Известно, что аминокислотный остаток аргинин(Arg), ближайший к олигопептиду SLWDQ с Nтерминального конца, и 67-SFLTKGP-73 играют важную роль при присоединении CD4 к gp120(Kwong, P.D., et al., Nature, vol. 398, 648-659,1998). Также известно, что несколько аминокислотных остатков, расположенных рядом с гомологической областью (289-KTIIVQLNETVKINCIRPNNKT-310), показанной на фиг. 16b сN-терминального конца, являются одной из об 004002 26 ластей, играющих важную роль при распознавании белка CD4 с помощью белка gp120(Kwong, P.D., et al., Nature, vol. 398, 648-659,1998). Таким образом, даже если о связывании между gp120 и CD4 неизвестно, оно может быть предсказано при помощи вышеописанного метода прогнозирования. Пример 11. CED-4/MAC-1. Нематода Caenorhabditis elegans - первый многоклеточный организм, генетический код которого был полностью расшифрован. Здесь приводится один пример, связанный с С.elegans. Белок CED-4 играет ключевую роль в контроле запрограммированной клеточной смерти. Было установлено, что белок МАС-1 прикрепляется кCED-4 и подавляет клеточную смерть (Wu et al.,Development, vol. 126, 9, 2021-2031, 1999). Таким образом, с целью подтверждения настоящего изобретения были изучены олигопептиды,составляющие часть данных двух белков, хотя связывание между МАС-1 и CED-4 известно. В результате, было обнаружено, что МАС-1 иCED-4 содержат в себе один и тот же олигопептид FPSVE, длина аминокислотной последовательности которого составляет 5 остатков, при этом настоящее изобретение было подтверждено. Индекс для данного олигопептида, рассчитанный из частоты встречаемости аминокислот в геноме С.elegans, составил 5,436. Помимо этого, как демонстрируется на фиг. 17, у данных двух белков имеется много гомологических областей, что настоятельно указывает на способность к связыванию этих белков (верхняя последовательность, CED-4; нижняя последовательность, МАС-1). Пример 12. АРР/BASE. АРР (белок-предшественник амилоида),который является одним из белков, вызывающих болезнь Альцгеймера, образует амилоид после расщепления на двух участках. Недавно был открыт фермент (BASE, бата секретаза),который расщепляет участок с аминотерминального конца двух участков расщепления(VASSAR et al., Science, 286(5440), 735-741,1999). Расщепление АРР энзимом BASE указывает на наличие взаимодействия данных двух белков. Для подтверждения настоящего изобретения были изучены олигопептиды, составляющие часть двух данных белков. АРР и BASE имеют гомологические олигопептиды WYFDV и WYYEV с аминокислотной последовательностью длиной 5 остатков, которые составляют часть каждого из них. Олигопептид WYFDV существует только в белке АРР из человеческих белков по SWISS-PROT версии 35. Человеческий белок, включающий в себя WYYEV, пока не зарегистрирован. Оба олигопептида обладают высокой специфичностью. Данный результат подтвердил настоящее изобретение. На фиг. 18 демонстрируются гомологические области двух данных белков (верхняя последовательность,АРР; нижняя последовательность, BASE). 27 Пример 13. Фурин и фактор фон Виллебранда. Фурин представляет собой внутриклеточную серии протеазу и связан с трактом выделительной системы, как фактор фон Виллебранда(VWF), альбумин и дополнительный С 3. Здесь приводится пример взаимодействия фурина сVWF. VWF отщепляется от белка-предшественника фурином, чтобы далее действовать в качестве зрелого белка. Отщепление фурином белкапредшественника VWF требует взаимодействия двух данных белков. Кроме того, фурин сам по себе при отщеплении становится из белкапредшественника фурина зрелым белком, чтобы действовать в качестве протеазы. Таким образом, чтобы подтвердить настоящее изобретение,был исследован олигопептид, составляющий часть белка-предшественника фурина и белкапредшественника VWF. Два этих белка содержат один и тот же олигопептид HCPPG в позициях 613-617 белка-предшественника фурина и в позициях 1176-1180 белка-предшественникаVWF (фиг. 19 а). Оба указанных местоположения находятся в пределах области зрелых белков. Олигопептид HCPPG присутствует только в белке-предшественнике фурина и белкепредшественнике VWF из человеческих белков по SWISS-PROT версии 35 и обладает высокой специфичностью с точки зрения частоты встречаемости. Белок-предшественник VWF расщепляется фурином на участке между 763-м аминокислотным остатком и 764-м аминокислотным остатком. Локальная линейная сверка сходства между белком-предшественником фурина и белком-предшественником VWF обнаруживает,что в области, расположенной рядом с участком расщепления фурином белка-предшественникаVWF, имеется часть области, гомологическая белку-предшественнику фурина (фиг. 19b). Таким образом, даже если предполагалось, что неизвестный белок является протеазой по мотиву активной части, а дополняющий белок, как и участок расщепления в дополняющем белке,неизвестны, настоящее изобретение позволяет спрогнозировать дополняющий белок, а также участок расщепления в дополняющем белке. Пример 14. АРР и РС 7. АРР альфа представляет собой пептид, образованный расщеплением белка-предшественника амилоида (АРР) на участке, отличном от двух участков расщепления для образования амилоида. Недавно было обнаружено, что РС 7(субтилизин/кексин конвертазы пробелка тип 7) участвует в расщеплении при образовании АРР альфа (Lopez-Perez E. et al., J. Neurochem., vol. 73, 5, 2056-2062, 1999). При изучении олигопептида, составляющего часть двух белков АРР и РС 7, выявлено, что АРР и РС 7 имеют гомологические олигопептиды DSDPSG и DSDPNG с длиной аминокислотной последовательности в 6 остатков, составляющих часть каждого из них. Олигопептид DSDPSG присутствует только в 28 белке АРР из человеческих белков по SWISSPROT версии 35. Человеческий белок, содержащий DSDPNG, пока не зарегистрирован. Оба олигопептида обладают весьма высокой специфичностью. Данный результат подтвердил настоящее изобретение. На фиг. 20 демонстрируются области, гомологические у двух данных белков. Между К и L 687-KLVFFAEDVGS-697 белка АРР на фиг. 20 расположен участок расщепления с образованием АРР альфа; на фиг. 20 демонстрируется, что гомологический этому участок последовательности (359-RMPFYAEECAS369) существует в РС 7. То есть данный пример показывает, что настоящее изобретение позволяет спрогнозировать белок, участвующий в расщеплении другого белка. Промышленная применимость Как было подробно описано выше, настоящее изобретение позволяет с использованием белковой базы данных спрогнозировать дополняющий белок, который взаимодействует с белком неизвестной функции, полученным путем генного анализа или с-ДНК анализа, или с белком известной функции. А именно, межбелковое взаимодействие в одном организме, генетический код которого расшифрован, может быть спрогнозировано на компьютере с использованием белковой базы данных на основе генного анализа или анализа с-ДНК, которые были недавно усовершенствованы. Если становится возможным компьютерное прогнозирование, то информация о белках, которые по прогнозу будут взаимодействовать между собой, может быть легко получена на основании компьютерного прогноза без применения рискованных методик, в которых результат зависит от использованной с-ДНК библиотеки, как, например, двугибридный метод. Такое прогнозирование стало возможным, и таким образом появилась возможность легко спрогнозировать последовательность олигопептидов, участвующих во взаимодействии, а также смоделировать на основе этой информации новое соединение, способное контролировать межбелковые взаимодействия. Настоящее изобретение позволяет эффективно расшифровывать межбелковые взаимодействия и может найти широкое применение в различных отраслях, включая биохимию, молекулярную биологию, фармацевтические разработки, сельское хозяйство и биотехнологии. В особенности, в фармацевтических разработках данное изобретение позволяет спрогнозировать механизм заболевания, который до сих пор не был известен, и дает возможность создания новых лекарственных препаратов. ФОРМУЛА ИЗОБРЕТЕНИЯ 1. Способ прогнозирования белка или полипептида, взаимодействующих с конкретным белком или полипептидом (А), который включает следующие стадии: 1) декомпозиция-перегруппировка последовательности аминокислот белка или полипептида (А) в серию олигопептидов, обладающих заданной длиной в качестве информации о последовательности; 2) поиск в базе данных по последовательностям аминокислот такого белка или полипептида (С), который включал бы в себя последовательность аминокислот для каждого члена данной серии или для белка или полипептида (D),содержащего последовательность аминокислот,гомологную последовательности аминокислот каждого члена данной серии; 3) выполнение локальной линейной сверки сходства указанного белка или полипептида (А) с отобранным белком или полипептидом (С) или с отобранным белком или полипептидом(D) и 4) прогнозирование того, является ли установленный белок или полипептид (С) и/или белок или полипептид (D) белком или полипептидом (В), который взаимодействует с белком или полипептидом (А), на основе результатов локальной линейной сверки последовательностей аминокислот и на основе значения, подсчитанного, исходя из частоты встречаемости аминокислот и /или частоты встречаемости вышеуказанных олигопептидов в вышеупомянутой базе данных по последовательностям аминокислот. 2. Способ по п.1, где олигопептид состоит из 4-14 аминокислот. 3. Среда для записи, несущая программу для прогнозирования белка или полипептида(В), который взаимодействует с конкретным белком или полипептидом (А), при этом данная среда для записи характеризуется включением вa) средство для ввода информации о последовательности аминокислот в белке или полипептиде (С) и запоминания этой информации;b) средство перегруппировки-декомпозиции вышеуказанной информации в серию олигопептидов, имеющих заданную длину в качестве информации о последовательности, и соответственно средство для запоминания полученной информации о последовательности;c) средство хранения введенной базы данных по белкам;d) средство для доступа к хранимой базе данных по белкам и для установления белка или полипептида (С), имеющего аминокислотную последовательность вышеуказанного олигопептида, или белка или полипептида (D), имеющего аминокислотную последовательность, гомологическую аминокислотной последовательности вышеуказанного олигопептида, и средство хранения и подсчета установленного результата;e) средство для выполнения локальной линейной сверки вышеуказанного белка или полипептида (А) с установленным белком или полипептидом (С) или с белком или полипептидом(D) и средство для хранения и подсчета результата иf) средство для получения результирующего значения частоты встречаемости аминокислоты и/или частоты встречаемости вышеназванного олигопептида по базе данных по белкам с последующим указанием индекса для прогноза белок-белковых взаимодействий на основе этого результирующего значения и результирующего значения вышеупомянутой локальной линейной сверки и средство для запоминания и вывода на дисплей результата и соответственно установленного белка или полипептида (В), который взаимодействует с белком или полипептидомg) средство для ранжирования силы белокбелковых взаимодействий у установленных белков или полипептидов (В), основывающегося на индексах, подсчитанных на основе результирующего значения локальной линейной сверки и результирующего значения частоты встречаемости аминокислоты и/или частоты встречаемости олигопептида в базе данных по белкам для случая, когда существует более чем один белок или полипептид (В) из тех, что были установлены, и средство для запоминания и демонстрации данного результата. 5. Среда по п.3 или 4, дополнительно содержащая, по крайней мере, следующее средство:h) средство для демонстрации полной длины аминокислотных последовательностей вышеуказанного белка или полипептида (А) и вышеуказанного белка или полипептида (В), кото 31 рый был установлен, с последующим индикаторным обозначением месторасположения частичной последовательности, которая подлежит линейной сверке, в полнодлиннотной последовательности для случая, когда частичные последовательности аминокислот сверяются посредством локальной линейной сверки между белком или полипептидом (А) и белком или полипептидом (В). 6. Среда по любому из пп.3, 4 и 5, дополнительно включающая в свой состав, по крайней мере, следующее средство:i) средство для расчета модели пространственной структуры для случая, когда пространственная структура вышеуказанного белка или полипептида (А) или вышеуказанного белка или полипептида (В), который был установлен, является известной, или для случая, когда моделирование по гомологии дает возможность построить модель пространственной структуры, с последующим выводом на дисплей этой структуры частичных аминокислотных последовательностей, которые выверяются по пространственной структуре путем локальной линейной сверки белка или полипептида (А) и у белка или полипептида (В). 7. Среда по любому из пп.3-5 или 6, дополнительно включающая в свой состав, по крайней мере, следующее средство:j) средство классификации и запоминания белков в базе данных по белкам для того, чтобы сузить зону поиска. 8. Среда по любому из предшествующих пунктов, дополнительно включающая в свой состав, по крайней мере, следующее средство:k) средство для посерийного ввода каждого белка в базу данных по белкам, такого как вышеуказанный белок или полипептид (А). 9. Среда по любому из предшествующих пунктов, дополнительно включающая в свой состав, по крайней мере, следующее средство:l) средство для хранения базы данных по геномам. 10. Устройство для прогнозирования межбелковых взаимодействий, включающее в свой состав те средства, носителем которых является среда по пп.3-8 или 9. 11. Способ определения белков или полипептидов, способных взаимодействовать друг с другом, который включает идентификацию белка или полипептида (В), взаимодействующего,как это было спрогнозировано на основе применения способа по п.1 или 2, с конкретным белком или полипептидом (А), а затем дальнейшее экспериментальное подтверждение наличия взаимодействия между белком или полипептидом (А) и белком или полипептидом (В). 12. Способ определения белков или полипептидов, взаимодействующих друг с другом,который включает идентификацию белка или полипептида (В), взаимодействующего с конкретным белком или полипептидом (А) соглас 004002 32 но прогнозу, сделанному с применением устройства по п.10, а затем экспериментальное подтверждение наличия взаимодействия между белком и полипептидом (А) и белком или полипептидом (В). 13. Белок или полипептид, который установлен способом по п.11 или 12. 14. Способ скрининга соединения, контролирующего взаимодействие конкретного белка или полипептида (А) и белка или полипептида(В), в котором применяется способ по п.1 или 2. 15. Способ скрининга соединения, контролирующего взаимодействие конкретного белка или полипептида (А) и белка или полипептида(В), в котором применяется устройство по п.10. 16. Соединение, полученное способом по п.14 или 15. 17. Соединение, способное контролировать взаимодействие конкретного белка или полипептида (А) и белка или полипептида (В), которое получено путем конструирования фармакосредств (drug design) на основе информации о соединении, полученном способом по п.14 или 15. 18. Олигопептид, содержащий аминокислотную последовательность SEQ ID No:1, который способен контролировать взаимодействие веротоксина 2 (VTII) с Bcl-2. 19. Агент против гибели клеток, содержащий олигопептид, включающий аминокислотную последовательность SEQ ID No:1. 20. Олигопептид, включающий аминокислотную последовательность, гомологную последовательности аминокислот SEQ ID No:1,который способен контролировать взаимодействие VTII и Bcl-2. 21. Полипептид, содержащий аминокислотную последовательность такую же, как у олигопептида по п.18 или 20, который способен контролировать взаимодействие VTII и Bcl-2. 22. Способ скрининга, способного контролировать взаимодействие VTII и Bcl-2, в котором используется олигопептид по п.18 или 20 и/или полипептид по п.21. 23. Способ определения нуклеотидной последовательности в олигонуклеотиде, кодирующей олигопептид, который вовлечен во взаимодействие конкретного белка или полипептида (А) и белка или полипептида (В), где используется способ п.1 или 2 или устройство для прогнозирования согласно п.10. 24. Набор серии комбинаций человеческих белков, в отношении которых получен прогноз,что они взаимодействуют друг с другом, с применением способа по п.1 или 2 или с помощью устройства согласно п.10. 25. Способ селекции комбинации белков,взаимодействующих друг с другом, где упомянутое взаимодействие связано с заболеванием; причем указанный способ включает селективный отбор комбинации на основе информации о каком-либо известном белке, который может 33 иметь отношение к этому заболеванию, из набора серии комбинаций по п.24. 26. Набор серии комбинаций белков, которые взаимодействуют друг с другом, где данное взаимодействие относится к заболеванию; при этом каждый член вышеуказанной серии отбирается по способу согласно п.25. 27. Способ скрининга соединения, которое контролирует взаимодействие комбинации белков и/или двух белков, отбираемых из серии комбинаций по п.26. 28. Соединение, полученное способом по п.27. 34 29. Способ прогнозирования процессируемого участка белка, включающий прогнозирование взаимодействия конкретного белка с ферментом, расщепляющим данный белок, с применением способа по п.1 или 2 или с применением устройства по п.10. 30. Полипептид, содержащий аминокислотную последовательность, которая в соответствии с полученным прогнозом включает участок процессируемого белка, полученный способом п.29, и/или включает частичную последовательность, гомологическую этому процессируемому участку.
МПК / Метки
МПК: C07K 7/06, G06F 17/30, G01N 33/68
Метки: прогнозирования, способ, белок-белковых, взаимодействий
Код ссылки
<a href="https://eas.patents.su/21-4002-sposob-prognozirovaniya-belok-belkovyh-vzaimodejjstvijj.html" rel="bookmark" title="База патентов Евразийского Союза">Способ прогнозирования белок-белковых взаимодействий</a>
Предыдущий патент: Способ и система предоставления маркетинговых скидок на товары
Следующий патент: Способ и устройство для анализа обменных волн
Случайный патент: Стальная нить, патентированная в висмуте